永井 忠一 2020.4.19
\[ y(x,\mathbf{w}) = w_0 + w_1x + w_2x^2 + \cdots + w_Mx^M = \sum_{j=0}^{M}w_jx^j \]\( M \)は多項式の次数(order)、ベクトル\( \mathbf w \)は多項式の係数ベクトル
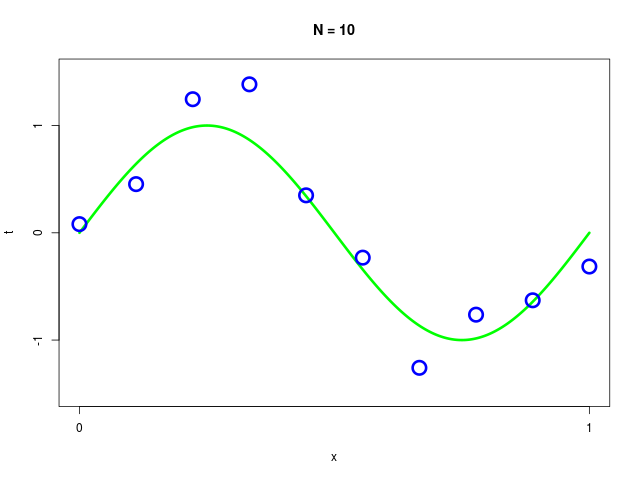
訓練データ集合(training data set)には、人工的に生成されたデータを使用

データを生成するプログラム
| R言語 |
|---|
|
R言語と開発環境(ESS)のインストール(私の環境ではapt)
| Linux |
|---|
|
ESS(Emacs Speaks Statistics)は、パッケージ名が ess から elpa-ess に変更になる。パッケージを『$ apt-cache search ess』などして探す。詳細は、『$ apt show elpa-ess』や『$ apt show ess』など
二乗和誤差\[ E(\mathbf{w}) = \frac{1}{2}\sum_{n=1}^N\{y(x_n,\mathbf{w}) - t_n\}^2 \]を最小化する
誤差関数 \( E(\mathbf w) \) を最小にする係数ベクトル \( \mathbf w \) を求める
|
\( \mathbf w \) の求め方\[ \frac{\partial E}{ \partial w_0} = \frac{\partial E}{ \partial w_1} = \cdots = \frac{\partial E}{ \partial w_M} = 0 \]を解く 2次式(\( M=2 \))の例。目的関数を書き下す\[ E(\mathbf w) = \frac{1}{2}\sum_{n}\{y(x_n,\mathbf{w}) - t_n\}^2 = \frac{1}{2}\sum_{n}(w_0 + w_1x_n + w_2x_n^2 - t_n)^2 \] 未知ベクトル \( \mathbf w \) で偏微分して \( 0 \) とおく\[ \begin{align} \frac{\partial E}{ \partial w_0} &= \frac{1}{2}\sum_{n}2(w_0 + w_1x_n + w_2x_n^2 - t_n) = \sum_{n}(w_0 + w_1x_n + w_2x_n^2 - t_n) = \left( \sum_n 1 \right)w_0 + \left( \sum_n x_n \right)w_1 + \left( \sum_n x_n^2 \right)w_2 - \sum_n t_n = 0 \\ ~~~ \frac{\partial E}{ \partial w_1} &= \frac{1}{2}\sum_{n}2x_n(w_0 + w_1x_n + w_2x_n^2 - t_n) = \sum_{n}x_n(w_0 + w_1x_n + w_2x_n^2 - t_n) = \left( \sum_n x_n \right)w_0 + \left( \sum_n x_n^2 \right)w_1 + \left( \sum_n x_n^3 \right)w_2 - \sum_n x_nt_n = 0 ~~~ \\ ~~~ \frac{\partial E}{ \partial w_2} &= \frac{1}{2}\sum_{n}2x_n^2(w_0 + w_1x_n + w_2x_n^2 - t_n) = \sum_{n}x_n^2(w_0 + w_1x_n + w_2x_n^2 - t_n) = \left( \sum_n x_n^2 \right)w_0 + \left( \sum_n x_n^3 \right)w_1 + \left( \sum_n x_n^4 \right)w_2 - \sum_n x_n^2t_n = 0 ~~~ \end{align} \] 行列の形で整理する\[ \begin{align} \begin{pmatrix} \sum_n 1 & \sum_n x_n & \sum_n x_n^2 \\ \sum_n x_n & \sum_n x_n^2 & \sum_n x_n^3 \\ \sum_n x_n^2 & \sum_n x_n^3 & \sum_n x_n^4 \end{pmatrix}\begin{pmatrix} w_0 \\ w_1 \\ w_2 \end{pmatrix} - \begin{pmatrix} \sum_n t_n \\ \sum_n x_nt_n \\ \sum_n x_n^2t_n \end{pmatrix} &= \mathbf 0 \\ \begin{pmatrix} \sum_n 1 & \sum_n x_n & \sum_n x_n^2 \\ \sum_n x_n & \sum_n x_n^2 & \sum_n x_n^3 \\ \sum_n x_n^2 & \sum_n x_n^3 & \sum_n x_n^4 \end{pmatrix}\begin{pmatrix} w_0 \\ w_1 \\ w_2 \end{pmatrix} &= \begin{pmatrix} \sum_n t_n \\ \sum_n x_nt_n \\ \sum_n x_n^2t_n \end{pmatrix} \\ \begin{pmatrix} w_0 \\ w_1 \\ w_2 \end{pmatrix} &= \begin{pmatrix} \sum_n 1 & \sum_n x_n & \sum_n x_n^2 \\ \sum_n x_n & \sum_n x_n^2 & \sum_n x_n^3 \\ \sum_n x_n^2 & \sum_n x_n^3 & \sum_n x_n^4 \end{pmatrix}^{-1}\begin{pmatrix} \sum_n t_n \\ \sum_n x_nt_n \\ \sum_n x_n^2t_n \end{pmatrix} \end{align} \] \( M \) 次式の場合(一般解)\[ \begin{align} \mathbf{A}\mathbf{w} &= \mathbf b \\ \mathbf{w} &= \mathbf{A}^{-1}\mathbf b \end{align} \]ただし、\[ A_{ij} = \sum_n x_n^{i+j},\ b_i = \sum_n x_n^i t_n \] \[ (i,j = 0, \ldots, M) \]行列・ベクトルの定義\[ \mathbf A \equiv \begin{pmatrix} \sum_n x_n^{0+0} & \cdots & \sum_n x_n^{0+M} \\ \vdots & \ddots & \vdots \\ \sum_n x_n^{M+0} & \cdots & \sum_n x_n^{M+M} \end{pmatrix},\ \mathbf b \equiv \begin{pmatrix} \sum_n x_n^0 t_n \\ \vdots \\ \sum_n x_n^M t_n \end{pmatrix},\ \mathbf w \equiv \begin{pmatrix} w_0 \\ \vdots \\ w_M \end{pmatrix} \] |
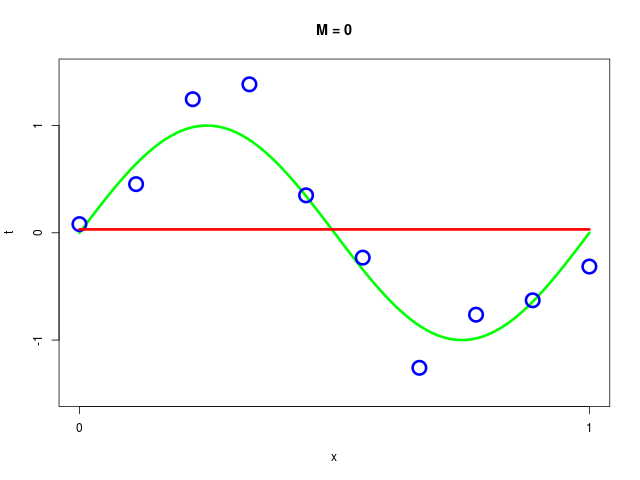
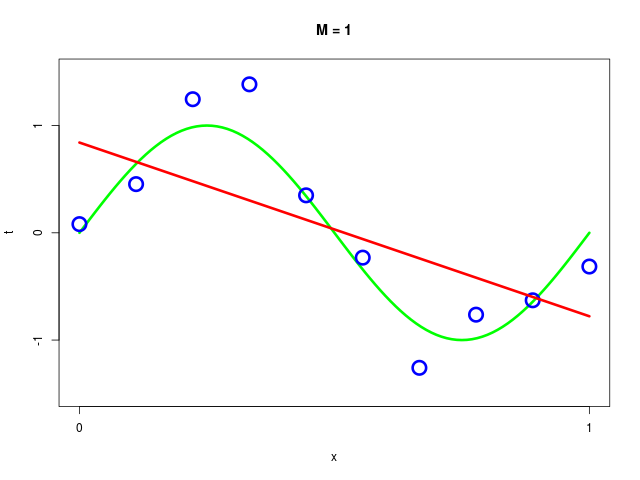
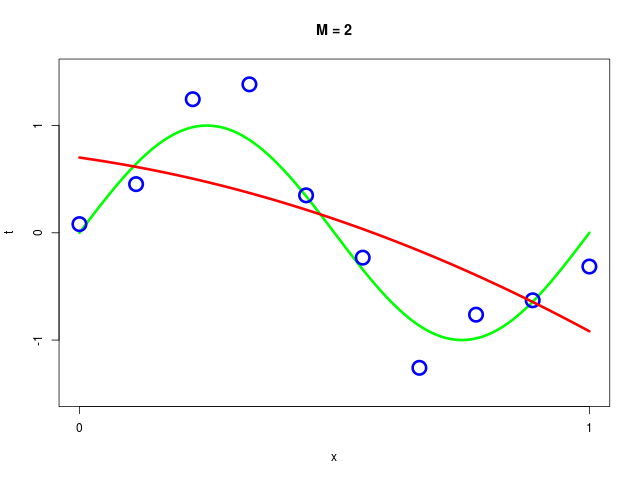
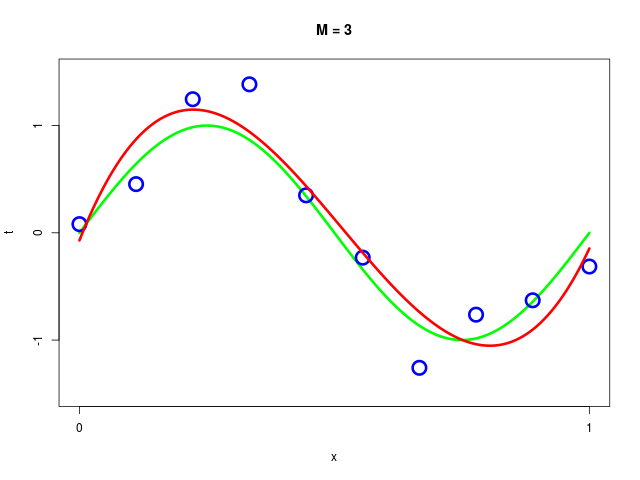
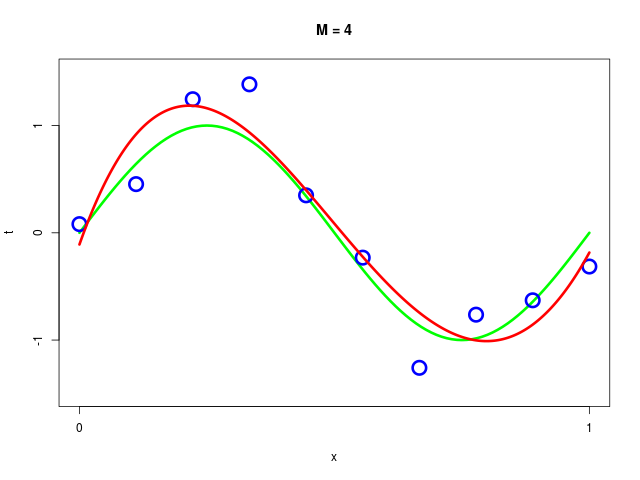
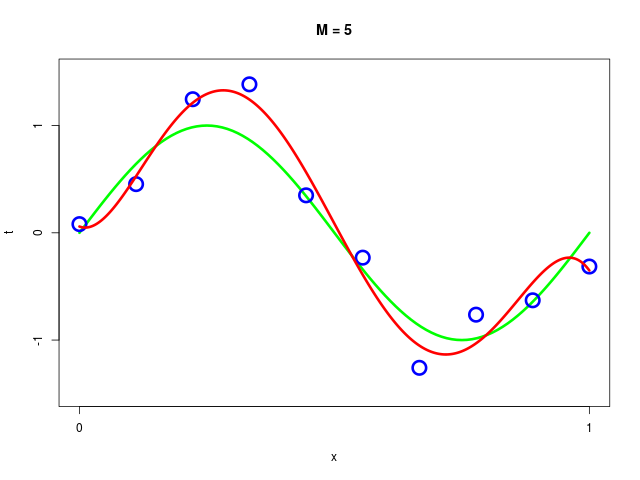
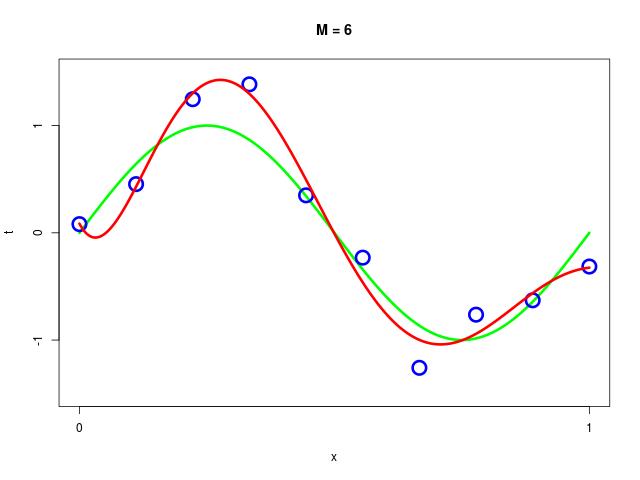
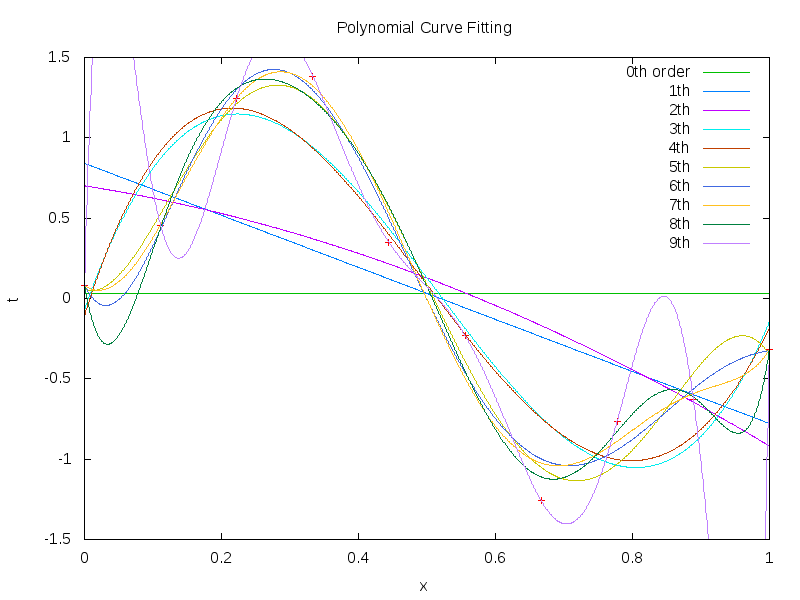
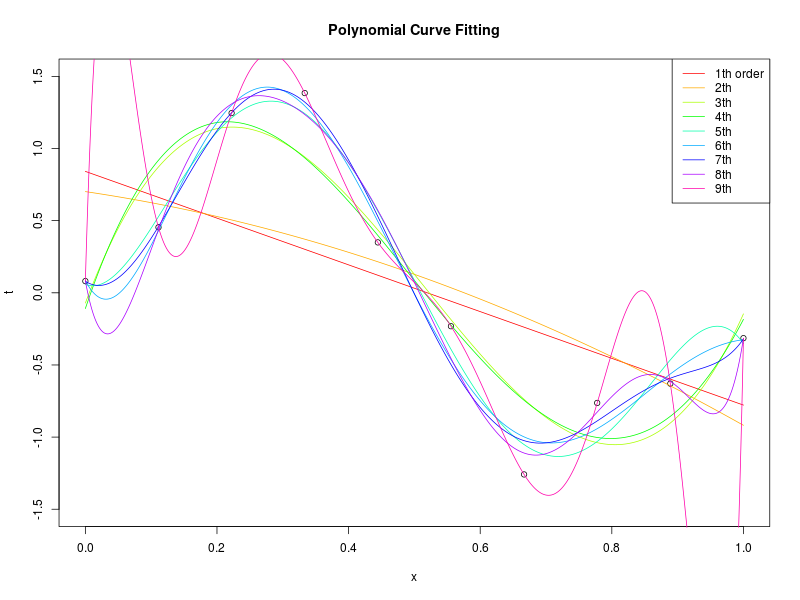
データ集合に次数 \( M = 1, 2, \ldots , 9\) を持つ多項式をフィッティングした結果
 |  |
 |  |
 |  |
 | 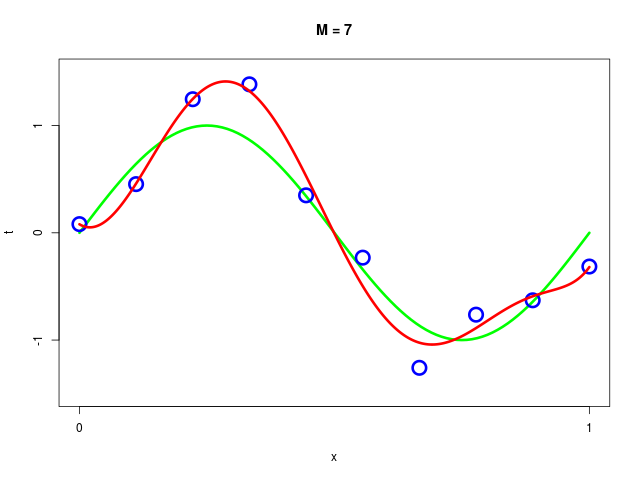 |
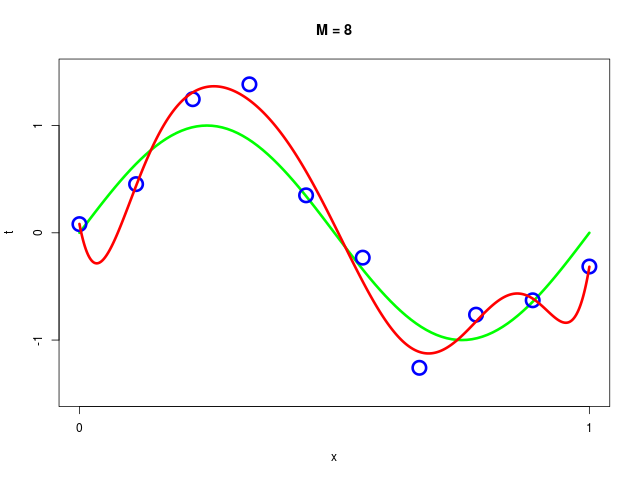 | 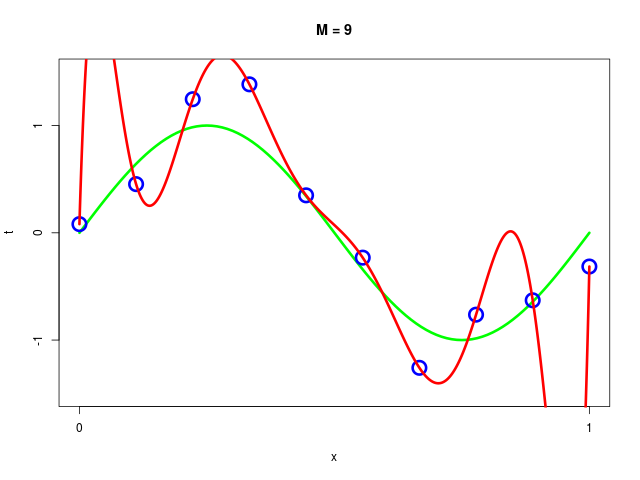 |
得られた解 \( \mathbf{w^*} \) の値を確認
| \( w^*_0 \) | \( w^*_1 \) | \( w^*_2 \) | \( w^*_3 \) | \( w^*_4 \) | \( w^*_5 \) | \( w^*_6 \) | \( w^*_7 \) | \( w^*_8 \) | \( w^*_9 \) | |
|---|---|---|---|---|---|---|---|---|---|---|
| \( M=0 \) | 0.03 | |||||||||
| \( M=1 \) | 0.84 | -1.62 | ||||||||
| \( M=2 \) | 0.70 | -0.67 | -0.95 | |||||||
| \( M=3 \) | -0.07 | 12.04 | -34.46 | 22.34 | ||||||
| \( M=4 \) | -0.11 | 13.47 | -41.66 | 33.90 | -5.78 | |||||
| \( M=5 \) | 0.06 | -1.79 | 85.53 | -325.65 | 406.34 | -164.85 | ||||
| \( M=6 \) | 0.09 | -8.90 | 173.27 | -703.27 | 1136.08 | -813.97 | 216.37 | |||
| \( M=7 \) | 0.08 | -2.85 | 73.40 | -111.04 | -538.48 | 1628.03 | -1555.80 | 506.34 | ||
| \( M=8 \) | 0.08 | -24.81 | 516.20 | -3450.90 | 12060.96 | -24602.79 | 29046.84 | -18229.98 | 4684.08 | |
| \( M=9 \) | 0.08 | 159.24 | -3734.96 | 34841.09 | -167830.87 | 465492.35 | -773458.81 | 760154.62 | -407139.88 | 91516.81 |
プログラム
| R言語 |
|---|
|
特定のモデルの、データ集合のサイズが変化したときの振る舞い
 | 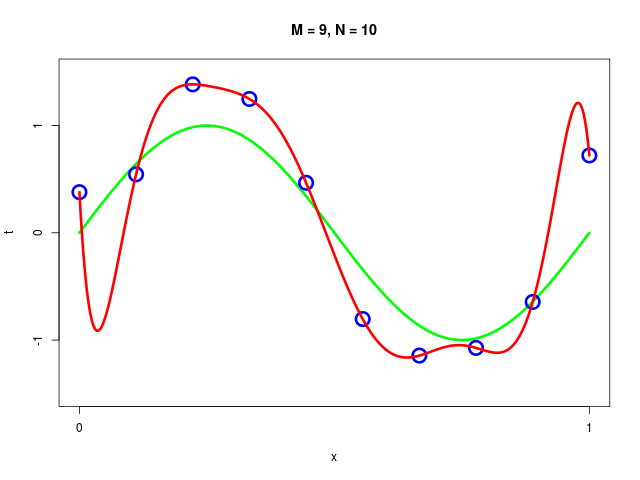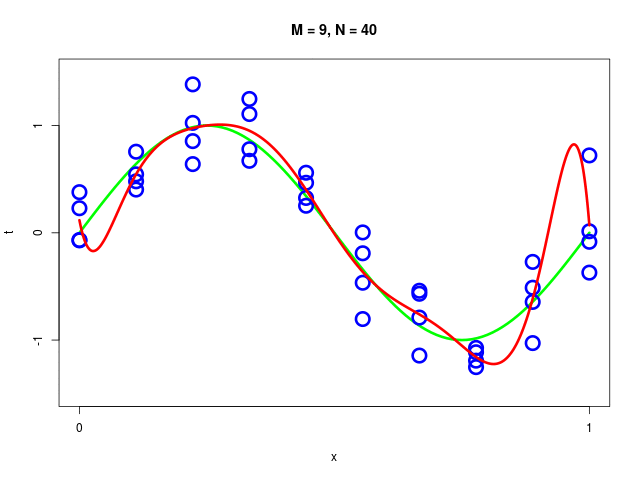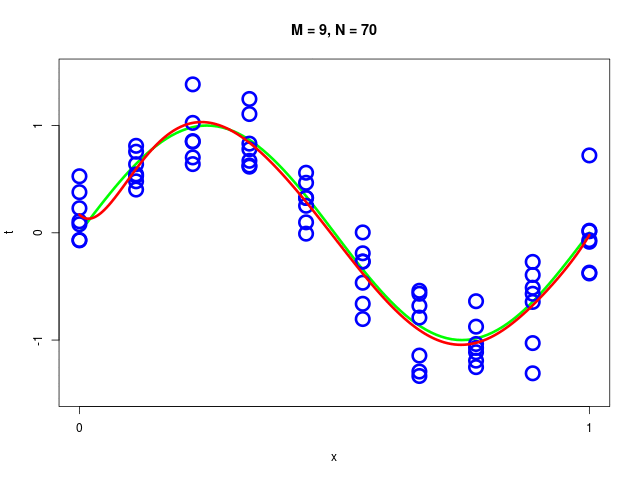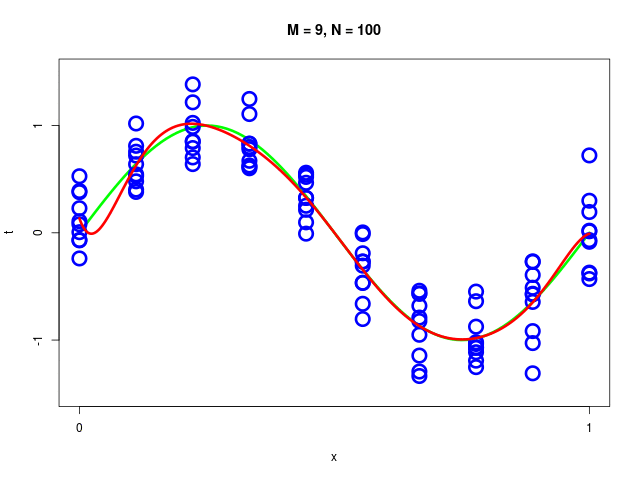 |
| R言語 |
|---|
|
| gnuplot |
|---|
|
|
| R言語 |
|---|
|
|
確率の基本的な法則
| 加法則(sum rule) | \[ ~ p(X)=\sum_Y p(X, Y) \] |
|---|---|
| 乗法則(product rule) | \[ ~ p(X,Y)=p(Y|X)p(X) \] |
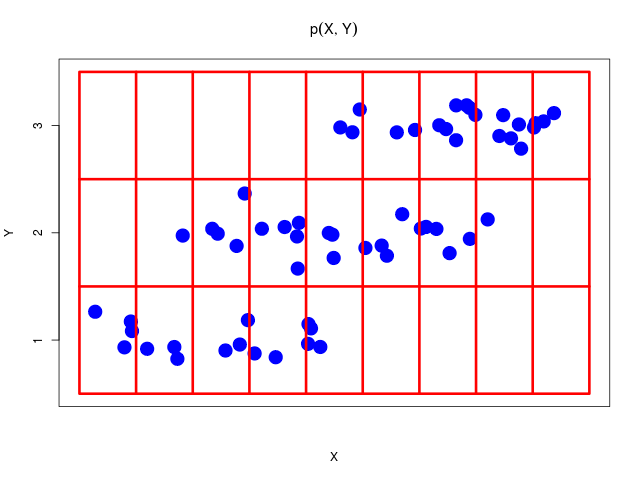
2つの確率変数\( X, Y \)を考える。\( X \)の値は\( \{x_i\}(i = 1,\ldots,M) \) 、\( Y \)の値は\( \{ y_j \}(j = 1,\ldots,L) \)の値をとる。総数\( N \)回の試行のうち、\( X = x_i\)かつ\( Y = y_j \)となる回数を\( n_{ij} \)、\( X = x_i \)となる回数を\( c_i \)、\( Y = y_j \)となる回数を\( r_j \)とする
同時確率(joint probability)\[ p(X=x_i, Y=y_j) = \frac{n_{ij}}{N} \]
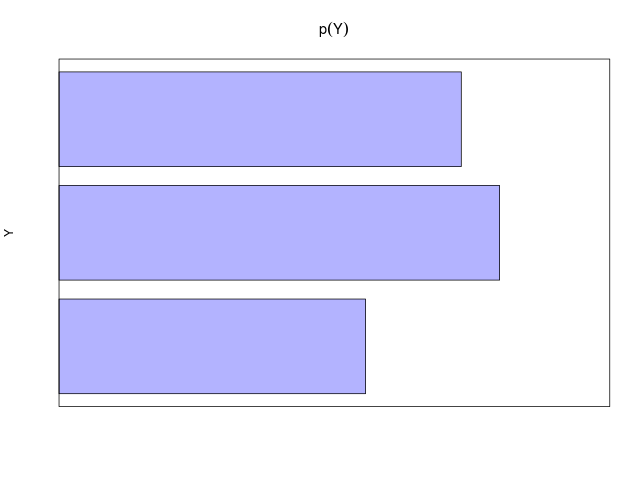
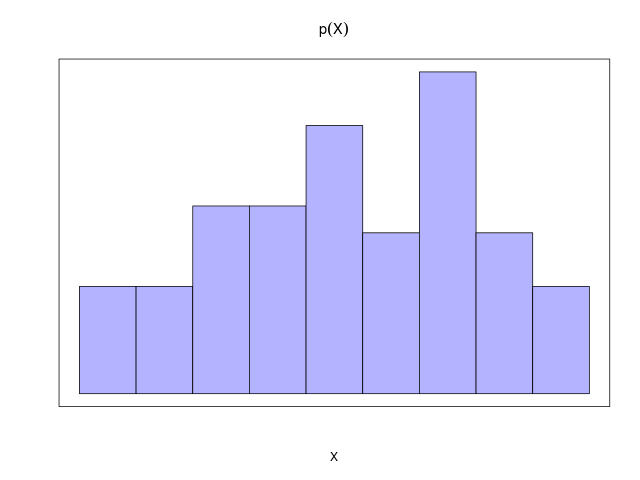
| (\( Y \)について和をとり、周辺化した場合) | \[ ~ p(X=x_i) = \frac{c_i}{N} = \sum_{j=1}^{L}p(X=x_i, Y=y_j) \] |
|---|---|
| (\( X \)について和をとり、周辺化した場合) | \[ ~ p(Y=y_j) = \frac{r_j}{N} = \sum_{i=1}^{M}p(X=x_i, Y=y_j) \] |
条件付き確率(conditional probability)
| \( X = x_i \)が与えられた\( Y = y_j \)の条件付き確率 | \[ ~ p(Y=y_j | X=x_i) = \frac{n_{ij}}{c_i} \] |
|---|---|
| \( Y = y_j \)が与えられた\( X = x_i \)の条件付き確率 | \[ ~ p(X=x_i | Y=y_j ) = \frac{n_{ij}}{r_j} \] |
上記の\( p(X=x_i, Y=y_j) = n_{ij}/N \)、\( p(X=x_i) = c_i/N \)および上式\( p(Y=y_j | X=x_i) = n_{ij}/c_i \)より\[ p(X=x_i, Y=y_j) = \frac{n_{ij}}{N} = \frac{n_{ij}}{c_i} \cdot \frac{c_i}{N} = p(Y=y_j | X=x_i)p(X=x_i) \]となる。これ(↑)が確率の乗法定理
確率変数\( X \)が値\( x_i \)をとる確率が\( p(X=x_i) \)。文脈上明らかなとき、確率変数\( X \)上の確率分布を\( p(X) \)、その分布が値\( x_i \)をとる確率を\( p(x_i) \)と簡易表記
 |  |
 | |
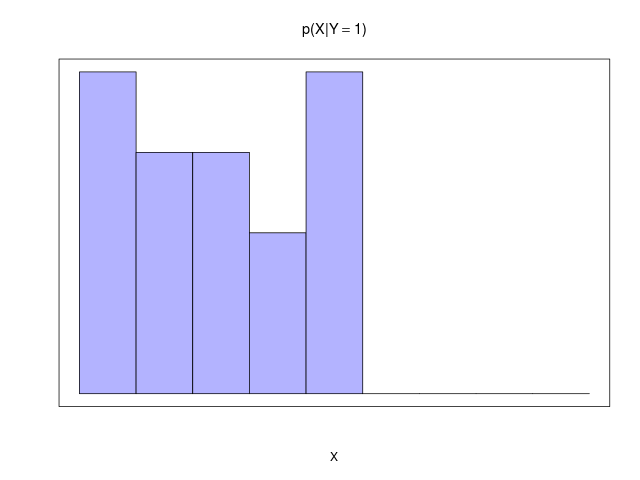 | |
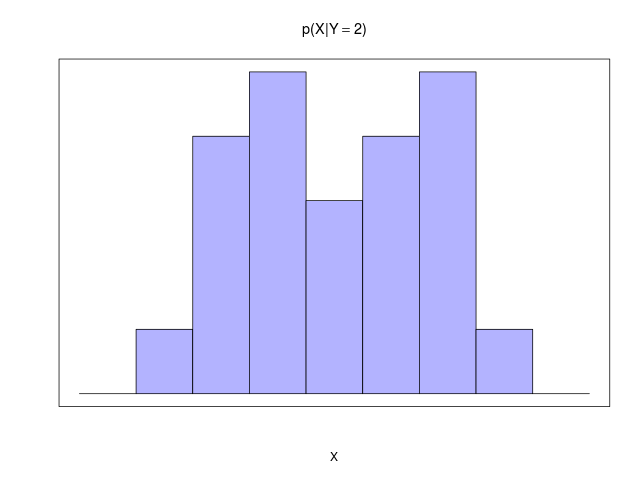 | |
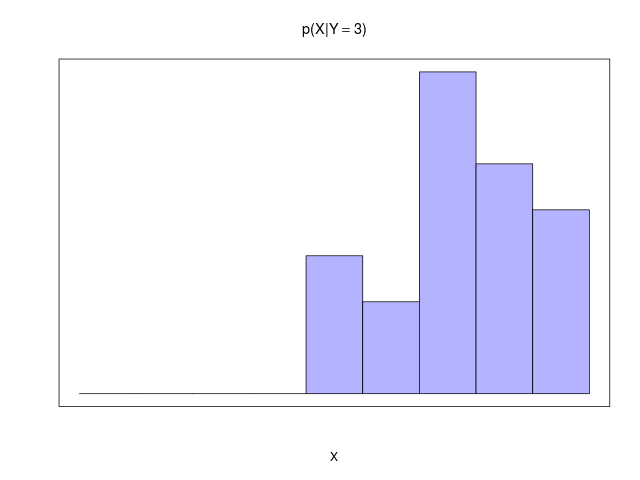 |
ソースコード
| R言語 |
|---|
|
乗法則の変形に過ぎないが、非常に有用な式変形の結果\[ p(Y | X) = \frac{p(X | Y)p(Y)}{p(X)} \](実質、条件付き確率の定義)
辺々\( p(X) \)を掛けて、乗法定理から導ける\[ \begin{align} p(Y | X) &= \frac{p(X | Y)p(Y)}{p(X)} \\ p(Y | X)p(X) &= p(X | Y)p(Y) \\ p(X, Y) &= p(Y, X) \end{align} \]対称性(symmetry property)\( p(X,Y) = p(Y,X) \)より両辺は等しい
ベイズの定理の分母の\( p(X) \)を\( Y \)について和をとった形、周辺確率\( p(X) = \sum_Y p(X | Y)p(Y) \)とみなして\[ p(Y | X) = \frac{p(X | Y)p(Y)}{\sum_Y p(X | Y)p(Y)} \]この形で書かれることも多い
(\( p(X) \)を、加法定理を使い\( p(X) = \sum_Y p(X, Y) \)とし、さらに乗法定理を使って\( p(X) = \sum_Y p(X | Y)p(Y) \)と書き換え、\[ p(Y | X) = \frac{p(X | Y)p(Y)}{p(X)} = \frac{p(X | Y)p(Y)}{\sum_Y p(X | Y)p(Y)} \]と変形)
ベイズの定理の分母は、分子に表れる量(\( p(X | Y)p(Y) \))で表すことができる。分母は、左辺の条件付確率をすべての\( Y \)について和をとったものが1になることを保証するための規格化(正規化)定数とみることができる(すべての事象の確率の和は1)
2変数の同時分布が周辺分布の積に分解できるとき\[ p(X, Y) = p(X)p(Y) \]\( X \)と\( Y \)は独立であるという。独立であるならば、乗法定理から\[ p(X|Y) = p(X),\ p(Y|X) = p(Y) \]であることがわかる
期待値
| 離散分布(discrete distribution) | 連続変数(continuous variable) |
|---|---|
| \[ \mathbb{E}[f] = \sum_x p(x)f(x) \] | \[ \mathbb{E}[f] = \int p(x)f(x)\,\mathrm dx \] |
多変数関数の期待値 \( \mathbb{E}[f] = \sum_{x,y}p(x,y)f(x,y) \)
どの変数について平均を取るのかを示すために添え字を使う
| 離散分布 | 連続変数 |
|---|---|
| \[ \begin{align} \mathbb{E}_x[f] &= \\ \mathbb{E}_x[f(x,y)] &= \sum_x p(x)f(x,y) \end{align} \] | \[ \begin{align} \mathbb{E}_x[f] &= \\ \mathbb{E}_x[f(x,y)] &= \int p(x)f(x,y)\,\mathrm dx \end{align} \] |
\( \mathbb{E}_x[f(x,y)] \)は、関数\( f(x,y) \)の\( x \)の分布に関する平均を表す。期待値は\( y \)の関数になることに注意
条件付分布(conditional distribution)についての条件付き期待値(conditional expectation)
| 離散分布 | 連続変数 |
|---|---|
| \[ \mathbb{E}_x[f|y] = \sum_x p(x|y)f(x,y) \] | \[ \mathbb{E}_x[f|y] = \int p(x|y)f(x,y)\,\mathrm dx \] |
分散(variance)の定義\[ \mathrm{var}[f] = \mathbb{E}\left[ (f(x) - \mathbb{E}[f(x)]^2 \right] \]
(分散の公式)\[ \mathrm{var}[f] = \mathbb{E}[f(x)^2] - \mathbb{E}[f(x)]^2 \]と書くこともできる
変数\( x \)自体の分散を\[ \mathrm{var} = \mathbb{E}[x^2] - \mathbb{E}[x]^2 \]によって与えられると考えることができる(\( f(x) = x \)の意味)
2つの確率変数 \( x \) と \( y \) の共分散(covariance)の定義\[ \begin{align} \mathrm{cov}[x,y] &= \mathbb{E}_{x,y}\left[ \{x - \mathbb{E}[x]\}\{y - \mathbb{E}[y]\} \right] \\ &= \mathbb{E}_{x,y}[xy] - \mathbb{E}[x]\mathbb{E}[y] \end{align} \]もし \( x \) と \( y \) が独立なら共分散は \( 0 \) になる
確率変数 \( \mathbf{x} \) と \(\mathbf{y} \) の2つのベクトルの場合、共分散は行列\[ \begin{align} \mathrm{cov}[\mathbf{x},\mathbf{y}] &= \mathbb{E}_{\mathbf{x},\mathbf{y}}\left[ \{\mathbf{x} - \mathbb{E}[\mathbf{x}]\}\{\mathbf{y}^{\mathrm T} - \mathbb{E}[\mathbf{y}^{\mathrm T}]\} \right] \\ &= \mathbb{E}_{\mathbf{x},\mathbf{y}}[\mathbf{x}\mathbf{y}^{\mathrm T}] - \mathbb{E}[\mathbf{x}]\mathbb{E}[\mathbf{y}^{\mathrm T}] \end{align} \]ベクトル \( \mathbf x \) の成分間の共分散は、シンプルな表記 \( \mathrm{cov}[\mathbf{x}] \equiv \mathrm{cov}[\mathbf{x},\mathbf{x}] \) を使う
\( x \)は確率変数、\( c \)が定数\[ \mathbb{E}[c] = c \]\[ \mathbb{E}[x + c] = \mathbb{E}[x] + c \]\[ \mathbb{E}[cx] = c\mathbb{E}[x] \]
\( x, y \)が確率変数\[ \mathbb{E}[x + y] = \mathbb{E}[x] + \mathbb{E}[y] \]
\( x \)は確率変数、\( c \)が定数\[ \mathrm{var}[c] = 0 \]\[ \mathrm{var}[x + c] = \mathrm{var}[x] \]\[ \mathrm{var}[cx] = c^2\mathrm{var}[x] \]
\( x, y \)は確率変数\[ \mathrm{var}[x + y] = \mathrm{var}[x] + \mathrm{var}[y] + 2\mathrm{cov}[x, y] \]\( x, y \)が独立ならば\[ \mathrm{var}[x + y] = \mathrm{var}[x] + \mathrm{var}[y] \]加法性が成り立つ(\(\ \mathrm{cov}[x, y] = 0 \) なので)
| ベルヌーイ分布 | 二値 | 離散 | パラメトリック(parametric) | |
| 二項分布 | ||||
| 多項分布 | 多値 | |||
| ガウス分布 | 1変数 | 連続 | ||
| 多次元ガウス分布 | 多変数 | |||
互いに排他なK個の状態の一つをとることができる離散変数
変数は、要素\( x_k \)の一つが\( 1 \)で、残りすべての要素が\( 0 \)の\( K \)次元ベクトル\( \mathbf{x} \)で表現される(→\( K \)種類の状態を表現)。たとえば、\( K = 6,\ x_3 = 1\)の場合(たとえば、サイコロの目の3が出た場合)\[ \mathbf{x} = (0, 0, 1, 0, 0, 0)^\mathrm{T} \]と表現される。このようなベクトル\( \mathbf{x} = (x_1, \ldots, x_K)^\mathrm{T} \)は\( \sum_{k=1}^K x_k = 1 \)を満たすことに注意
\( x_k = 1 \)となる確率をパラメータ\( \mu_k \)で表すと、\( \mathbf{x} \)の分布は\[ p(\mathbf{x}|\boldsymbol \mu) = \prod_{k=1}^{K} \mu_k^{x_k} = \begin{cases} \mu_1 & (x_1 = 1) \\ & \vdots \\ \mu_K & (x_K = 1) \end{cases} \]で与えられる
| サイコロの例: | \[ ~ p(\mathbf{x}|\boldsymbol \mu) = p\left((0,0,1,0,0,0)^\mathrm{T}|(\mu_1, \mu_2,\mu_3,\mu_4,\mu_5,\mu_6)^\mathrm{T}\right) = \mu_3 \] |
|---|
ただし、\( \boldsymbol \mu = (\mu_1, \ldots, \mu_K)^\mathrm{T} \)は確率を表すので\[ \mu_k \geq 0,\ \sum_k \mu_k = 1 \]に拘束される
正規分布(normal distribution)としても知られ、連続変数の分布に広く使用されているモデル
1変数の場合
\[ \mathcal{N}(x|\mu,\sigma^2) = \frac{1}{(2\pi\sigma^2)^{1/2}}\exp\left\{ -\frac{1}{2\sigma^2}(x - \mu)^2 \right\} \] |
\( \mu \):平均 |
確率密度の要件を満たす(\( x \) がどの値をとっても \( 0 \) より大きい(\( \exp \)なので)、積分は教科書など)\[ \mathcal{N}(x|\mu,\sigma^2) > 0,\ \int_{-\infty}^\infty \mathcal{N}(x|\mu,\sigma^2)\,\mathrm dx = 1 \](確率密度が満たすべき条件\[ p(x) \geq 0,\ \int p(x)\,\mathrm dx = 1 \]\( p(x) \) は \( 0 \) 以上で、(関数が定義されている)全区間で積分すると \( 1 \))
期待値と分散\[ \mathbb{E}[x] = \int_{-\infty}^\infty \mathcal{N}(x|\mu,\sigma^2)x\,\mathrm dx = \mu \]\[ \mathbb{E}[x^2] = \int_{-\infty}^\infty \mathcal{N}(x|\mu,\sigma^2)x^2\,\mathrm dx = \mu^2 + \sigma^2 \]\[ \mathrm{var}[x] = \mathbb{E}[x^2] - \mathbb{E}[x]^2 = \sigma^2 \](計算は教科書など)
ガウス分布は、複数の確率変数の和を考えているときにも生じる。(Laplaceによる)中心極限定理
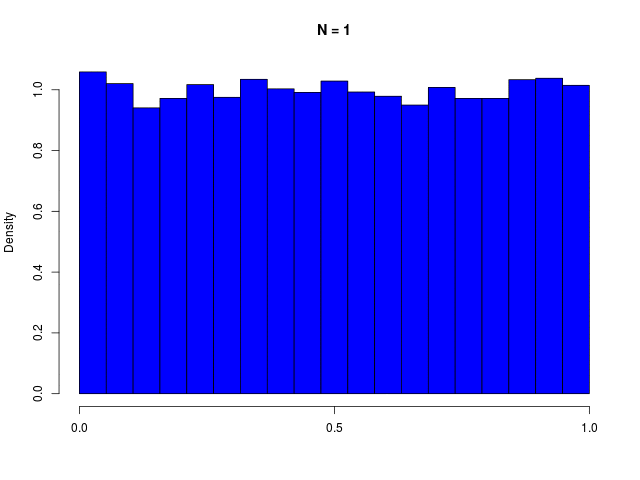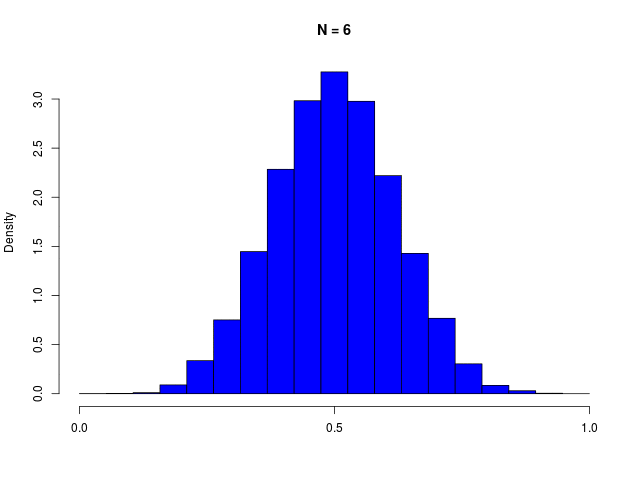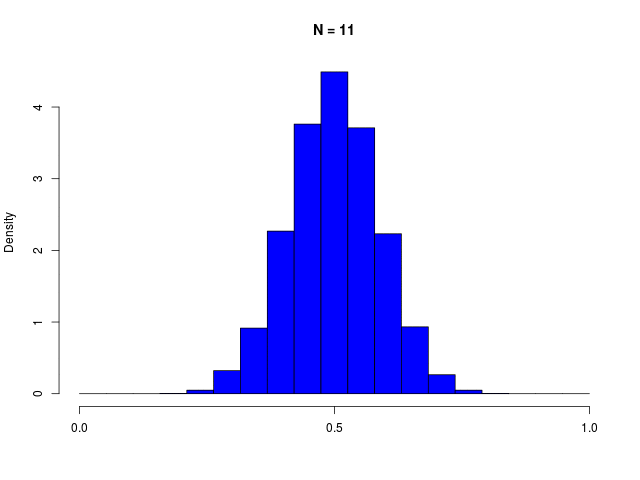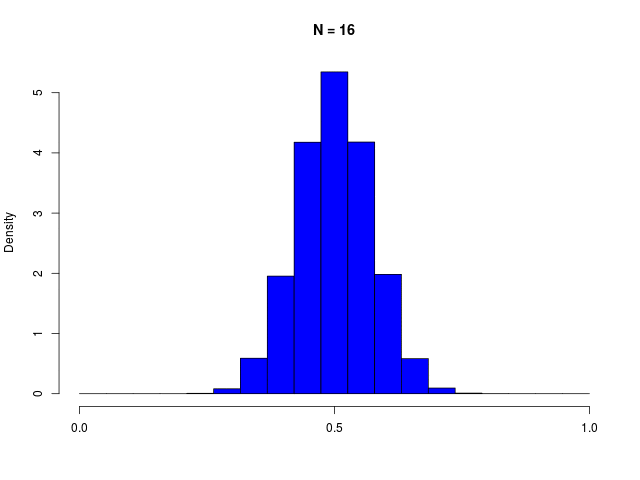 |
シミュレーションのソースコード
| R言語 |
|---|
|
二値確率変数の \( N \) 個の和で定義される \( m \) の分布である、二項分布(binomial distribution)は、\( N \to \infty \) でガウシアンに従う
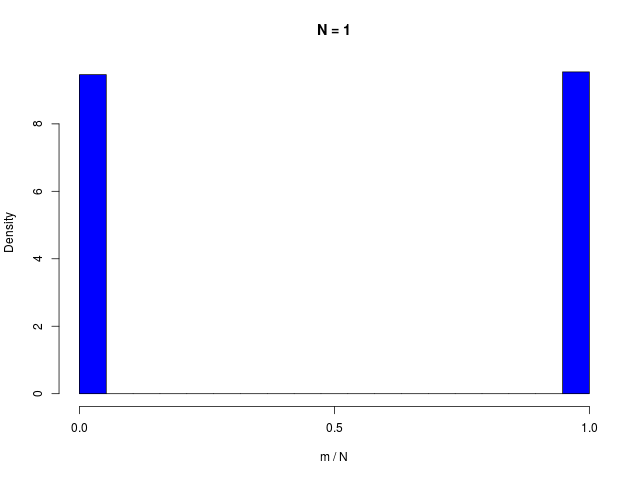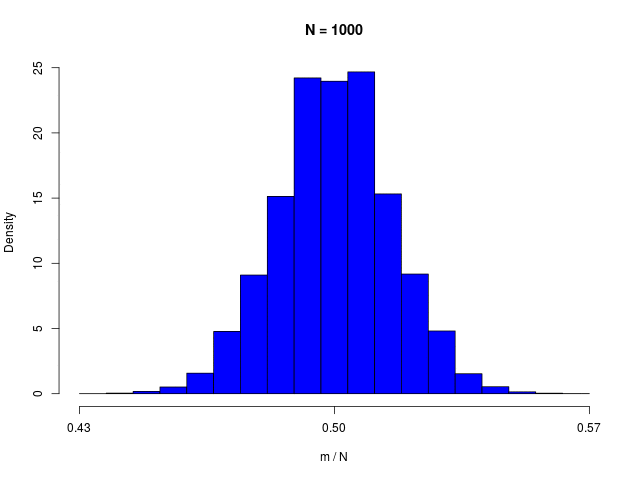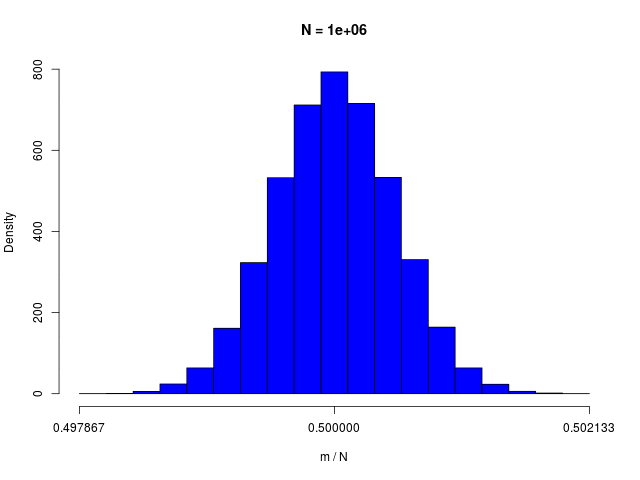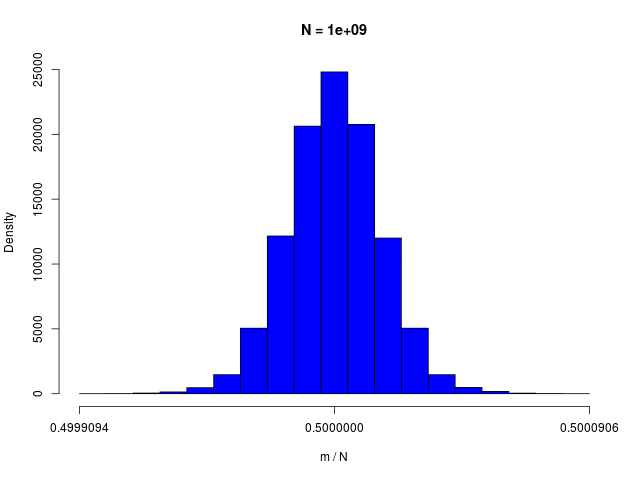 | 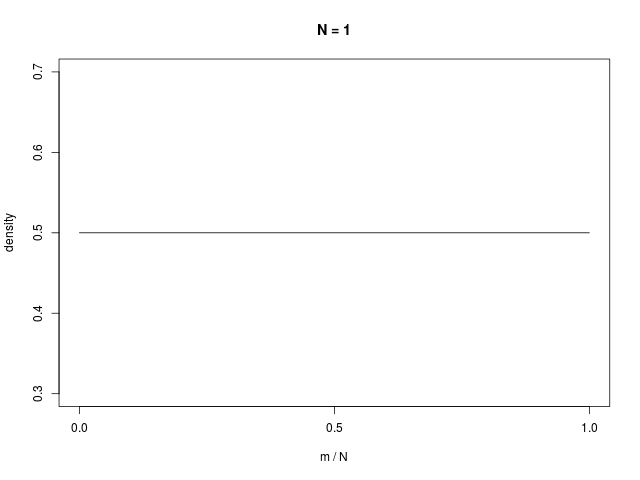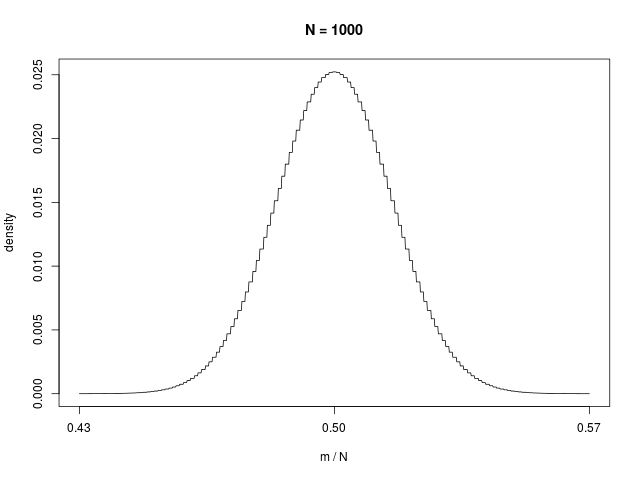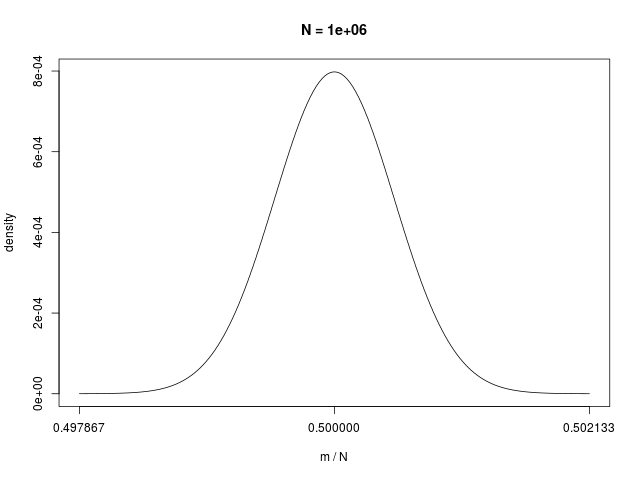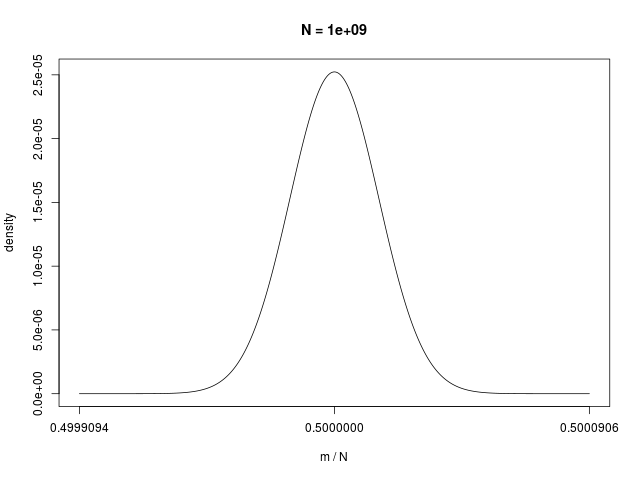 |
ソースコード
| R言語 |
|---|
|
|
\( D \) 次元ベクトル \( \mathbf x \) の多次元ガウス分布
\[ \mathcal{N}(\mathbf{x}|\boldsymbol \mu,\boldsymbol \Sigma) = \frac{1}{(2\pi)^{D/2}}\frac{1}{|\boldsymbol\Sigma|^{1/2}}\exp\left\{ -\frac{1}{2}(\mathbf x - \boldsymbol\mu)^\mathrm T\boldsymbol\Sigma^{-1}(\mathbf x - \boldsymbol\mu) \right\} \] | |
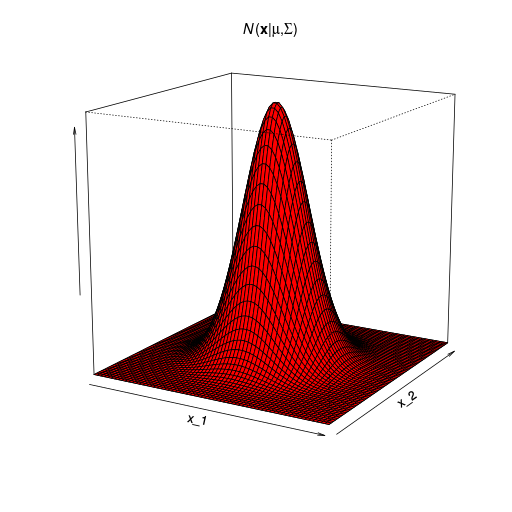 | \( \boldsymbol \mu \):\( D \)次元平均ベクトル |
\( D \)が1次元の場合
| cf. | \[ \mathcal{N}(x|\mu,\sigma^2) = \frac{1}{(2\pi\sigma^2)^{1/2}}\exp\left\{ -\frac{1}{2\sigma^2}(x - \mu)^2 \right\} = \frac{1}{(2\pi)^{1/2}}\frac{1}{(\sigma^2)^{1/2}}\exp\left\{ -\frac{1}{2}\frac{1}{\sigma^2}(x - \mu)^2 \right\} \] |
|---|---|
| \( \mu \):平均 \( \sigma^2 \):分散 | |
R言語のmvtnormパッケージのインストール(私の環境ではapt)
| Linux |
|---|
|
利用例のサンプルコード
| R言語 |
|---|
|
\( \mathbf x \)が多次元ガウス分布に従うとき\[ \mathbb E[\mathbf x] = \boldsymbol \mu \]\[ \mathbb E[\mathbf x \mathbf{x}^\mathrm T] = \boldsymbol \mu \boldsymbol{\mu}^\mathrm T + \boldsymbol \Sigma \]\[ \mathrm{cov}[\mathbf x] = \boldsymbol \Sigma \]となる(導出は教科書)
(\( \boldsymbol \Sigma \)の固有ベクトル\( \mathbf u_1, \ldots, \mathbf u_D \))
| 共分散行列が、一般(general form)の場合 | 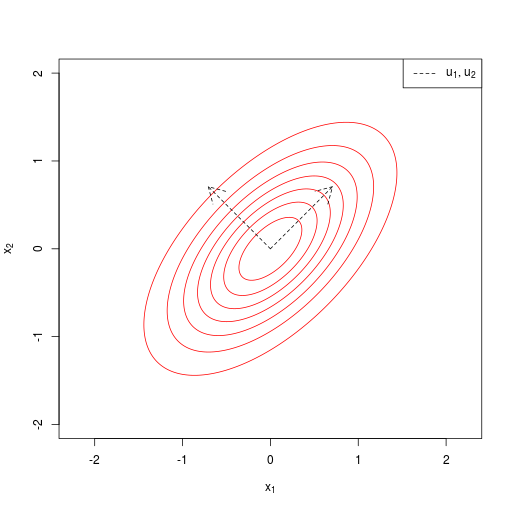 |
|---|---|
| 対角行列(diagonal) | 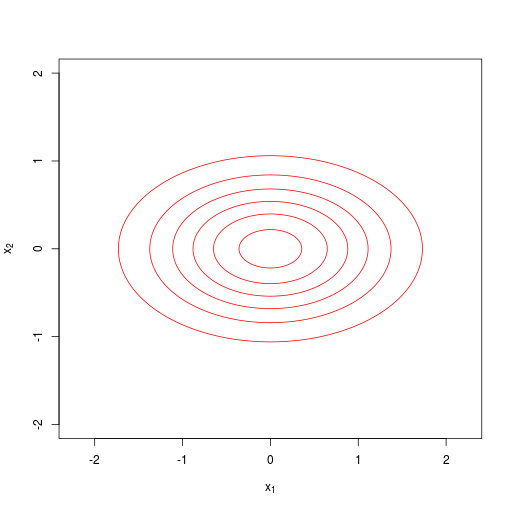 |
| \[ \boldsymbol \Sigma = \mathrm{diag}(\sigma^2_i) = \begin{pmatrix} \sigma^2_1 & & & 0 \\ & \sigma^2_2 & & \\ & & \ddots & \\ 0 & & & \sigma^2_D \end{pmatrix} \] | |
| 単位行列に比例(proportional to the identity matrix) | 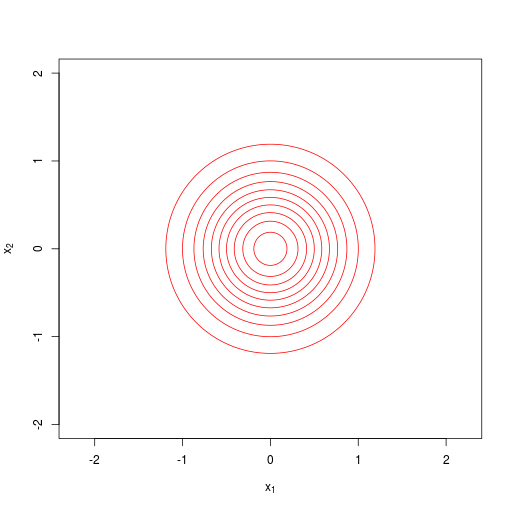 等方共分散行列(isotropic covariance matrices) (等方的なガウス分布) |
| \[ \boldsymbol \Sigma = \sigma^2 \mathbf{I} = \begin{pmatrix} \sigma^2 & & & 0 \\ & \sigma^2 & & \\ & & \ddots & \\ 0 & & & \sigma^2 \end{pmatrix} \] |
ソースコード
| R言語 |
|---|
|
(結論として、)多変数ガウス分布の重要な性質は、条件付きガウス分布も、周辺分布も、同様にガウス分布になる
同時ガウス分布 \( \mathcal{N}(\mathbf x|\boldsymbol \mu, \boldsymbol \Sigma) \)\[ \mathbf{x} = \begin{pmatrix} \mathbf x_a \\ \mathbf x_b \end{pmatrix},\ \boldsymbol \mu = \begin{pmatrix} \boldsymbol \mu_a \\ \boldsymbol \mu_b \end{pmatrix} \]\[ \boldsymbol \Sigma = \begin{pmatrix} \boldsymbol \Sigma_{aa} & \boldsymbol \Sigma_{ab} \\ \boldsymbol \Sigma_{ba} & \boldsymbol \Sigma_{bb} \end{pmatrix},\ \boldsymbol \Lambda = \begin{pmatrix} \boldsymbol \Lambda_{aa} & \boldsymbol \Lambda_{ab} \\ \boldsymbol \Lambda_{ba} & \boldsymbol \Lambda_{bb} \end{pmatrix} \equiv \boldsymbol \Sigma^{-1} \]
共分散行列の逆行列\[ \boldsymbol \Lambda \equiv \boldsymbol \Sigma^{-1} \]を精度行列(precision matrix)。ここで強調されるべきことは、\[ \begin{pmatrix} \boldsymbol \Lambda_{aa} & \boldsymbol \Lambda_{ab} \\ \boldsymbol \Lambda_{ba} & \boldsymbol \Lambda_{bb} \end{pmatrix} \equiv \begin{pmatrix} \boldsymbol \Sigma_{aa} & \boldsymbol \Sigma_{ab} \\ \boldsymbol \Sigma_{ba} & \boldsymbol \Sigma_{bb} \end{pmatrix}^{-1} \]たとえば、\( \boldsymbol \Lambda_{aa} \) は単純に \( \boldsymbol \Sigma_{aa} \) の逆行列によって与えられるわけではない
条件付き分布\[ p(\mathbf x_a|\mathbf x_b) = \mathcal N(\mathbf x_a|\boldsymbol\mu_{a|b}, \mathbf \Lambda_{aa}^{-1}) \]\[ \boldsymbol\mu_{a|b} = \boldsymbol\mu_a - \boldsymbol\Lambda_{aa}^{-1}\boldsymbol\Lambda_{ab}(\mathbf x_b - \boldsymbol\mu_b) \]
周辺分布\[ p(\mathbf x_a) = \mathcal N(\mathbf x_a|\boldsymbol\mu_a, \boldsymbol \Sigma_{aa}) \]
(導出は教科書)
ガウス周辺分布 \( p(\mathbf x) \) とガウス条件付き分布 \( p(\mathbf y|\mathbf x) \) が与えられていると仮定。周辺分布 \( p(\mathbf y)\) と条件付き分布 \( p(\mathbf x|\mathbf y) \) を求めたい\[ p(\mathbf{x}) = \mathcal N(\mathbf x|\boldsymbol\mu, \boldsymbol\Lambda^{-1}) \]\[ p(\mathbf y|\mathbf x) = \mathcal N(\mathbf y|\mathbf{A x} + \mathbf b,\mathbf L^{-1}) \]ただし、\( \boldsymbol\mu \)、\( \mathbf A \)、\( \mathbf b \) は平均を決定するパラメータ、\( \boldsymbol\Lambda \) と \( \mathbf L \) は精度行列
(一般解)
\( \mathbf y \) の周辺分布と \( \mathbf y \) が与えられた \( \mathbf x \) の条件付き分布\[ p(\mathbf y) = \mathcal N(\mathbf y|\mathbf A\boldsymbol\mu + \mathbf b, \mathbf L^{-1} + \mathbf A\boldsymbol\Lambda^{-1}\mathbf A^\mathrm T) \]\[ p(\mathbf x|\mathbf y) = \mathcal N(\mathbf x|\boldsymbol\Sigma\{ \mathbf A^\mathrm T\mathbf L(\mathbf y - \mathbf b) + \boldsymbol{\Lambda \mu} \}, \boldsymbol\Sigma) \]
ただし\[ \boldsymbol\Sigma = (\mathbf\Lambda + \mathbf A^\mathrm T\mathbf L\mathbf A)^{-1} \]
(導出は教科書)
(パラメータ推定)
観測値のデータ集合\[ \mathcal D = \{ x_1,\ldots,x_N \} \]これらが、ガウス分布からそれぞれ独立に得られたとする(独立同分布(i.i.d.)を仮定)
尤度関数\[ p(\mathcal D|\mu,\sigma^2) = \prod_{n=1}^N\mathcal N(x_n|\mu,\sigma^2) = \prod_{n=1}^N\frac{1}{\sqrt{2\pi\sigma^2}}\exp\left\{ -\frac{1}{2\sigma^2}(x_n - \mu)^2 \right\} \]
(尤度、尤度関数。同時確率分布の式\[ \mathbf x = (x_1,\ldots,x_N)^\mathrm T,\ p(\mathbf x|\mu,\sigma^2) = \prod_{n=1}^N\mathcal N(x_n|\mu,\sigma^2) \]を、観測データ \( \mathbf x \) を固定して、(データの背後にある)確率分布のパラメタ \(\mu,\sigma^2\) の方を変数としてみる。(同じ式を、式の見方を変える。)尤度関数は確率ではないので、変数が取り得る全ての範囲を積分しても \( 1 \) にはならない。尤度関数の値(尤度)は、そのパラメタがもとの確率分布を表している尤もらしさ)
(尤度を最大化する(最も尤もらしい) \( \mu,\sigma^2 \) を求める)
対数尤度関数\[ \ln p(\mathcal D|\mu,\sigma^2) = -\frac{1}{2\sigma^2}\sum_{n=1}^N(x_n - \mu)^2 - \frac{N}{2}\ln \sigma^2 - \frac{N}{2}\ln 2\pi \]
対数尤度を最大化するパラメータを求める(対数尤度関数をパラメタで偏微分して \( 0 \) となるところ)\[ \frac{\partial}{\partial\mu}\ln p(\mathcal D|\mu,\sigma^2) = 0,\ \frac{\partial}{\partial\sigma^2}\ln p(\mathcal D|\mu,\sigma^2) = 0 \](計算省略)
最尤推定解
| \[ \mu_\mathrm{ML} = \frac{1}{N}\sum_{n=1}^N x_n ~ \] | ←サンプル平均 |
| \[ \sigma^2_\mathrm{ML} = \frac{1}{N}\sum_{n=1}^N(x_n - \mu_\mathrm{ML})^2 ~ \] | ←サンプル分散 |
\( D \) 次元の多次元ガウス分布の最尤推定解
|
尤度関数\[ ~~~ p(\mathbf X|\boldsymbol\mu, \boldsymbol\Sigma) = \prod_{n=1}^N\mathcal N(\mathbf x_n|\boldsymbol\mu, \mathbf\Sigma) = \prod_{n=1}^N\left[ \frac{1}{(2\pi)^\frac{D}{2}|\mathbf\Sigma|^{\frac{1}{2}}}\exp\left\{ -\frac{1}{2}(\mathbf x_n - \boldsymbol\mu)^\mathrm T\boldsymbol\Sigma^{-1}(\mathbf x_n - \boldsymbol\mu) \right\} \right] ~~~ \] 対数尤度関数\[ \ln p(\mathbf X|\boldsymbol\mu, \boldsymbol\Sigma) = -\frac{ND}{2}\ln(2\pi) - \frac{N}{2}\ln|\boldsymbol \Sigma| - \frac{1}{2}\sum_{n=1}^N(\mathbf x_n - \boldsymbol\mu)^\mathrm T\boldsymbol\Sigma^{-1}(\mathbf x_n - \mu) \] 対数尤度を最大化\[ \frac{\partial}{\partial\boldsymbol\mu}\ln p(\mathbf X|\boldsymbol\mu, \boldsymbol\Sigma) = 0,\ \frac{\partial}{\partial\boldsymbol\Sigma}\ln p(\mathbf X|\boldsymbol\mu, \boldsymbol\Sigma) = 0 \] 最尤推定解\[ \begin{align} \boldsymbol\mu_\mathrm{ML} &= \frac{1}{N}\sum_{n=1}^N\mathbf x_n \\ \boldsymbol\Sigma_\mathrm{ML} &= \frac{1}{N}\sum_{n=1}^N(\mathbf x_n - \boldsymbol\mu_\mathrm{ML})(\mathbf x_n - \boldsymbol\mu_\mathrm{ML})^\mathrm T \end{align} \](サンプル平均とサンプル共分散) |
ベイズ確率
ベイズの定理\[ p(\boldsymbol\theta|\mathcal D) = \frac{p(\mathcal D|\boldsymbol\theta)p(\boldsymbol\theta)}{p(\mathcal D)} \](ベイズの定理の分母 \( p(\mathcal D) \) は(規格化)定数)
\[ p(\boldsymbol\theta|\mathcal D) \propto p(\mathcal D|\boldsymbol\theta)p(\boldsymbol\theta) \](確率変数 \( \boldsymbol\theta \) は推定されるパラメタ、\( \mathcal D \) は観測データ。\( p(\boldsymbol\theta|\mathcal D) \) は、データ \( \mathcal D \) が観測されたという条件のもとでパラメタが \( \boldsymbol\theta \) となる確率。\( p(\mathcal D|\boldsymbol\theta) \) は尤度関数で、パラメタが \( \boldsymbol\theta \) であったとしたときにデータ \( \mathcal D \) が観測される尤もらしさ(同時確率を、\( \boldsymbol\theta \) を変数とみなす)。\( p(\boldsymbol\theta) \) は、どのようなデータ \( \mathcal D \) が観測されるかに関わらず、パラメタが \( \boldsymbol\theta \) となる確率)
(逐次推定(sequential estimation)と共役事前分布(conjugate prior)について)
(MAP(maximum a posteriori)推定(最大事後確率推定)について → 事後確率分布のモード(最頻値)を(予測値として)採用)
ガウス分布\[ \mathcal N(x|\mu,\sigma^2) = \frac{1}{\sqrt{2\pi\sigma^2}}\exp\left\{ -\frac{1}{2\sigma^2}(x - \mu)^2 \right\} \]は、平均 \( \mu \) と分散 \( \sigma^2 \) の2つのパラメタを持つため
平均 \( \mu \) の推定(分散 \( \sigma^2 \) は既知)
(たとえば、事前に実験するなどしてセンサーノイズの分散を決定してある、というような場合)
|
尤度関数\[ \begin{align} p(\mathbf x|\mu) &= \prod_{n=1}^N \mathcal N(x_n|\mu,\sigma^2) \\ &= \prod_{n=1}^N \frac{1}{(2\pi\sigma^2)^{1/2}}\exp\left\{ -\frac{1}{2\sigma^2}(x_n - \mu)^2 \right\} \\ &= \frac{1}{(2\sigma^2)^{N/2}}\exp\left\{ -\frac{1}{2\sigma^2}\sum_{n=1}^N(x_n - \mu)^2 \right\} \end{align} \] 共役事前分布(は、ガウス分布)\[ p(\mu) = \mathcal N(\mu|\mu_0,\sigma^2_0) = \frac{1}{(2\pi\sigma^2_0)^{1/2}}\exp\left\{ -\frac{1}{2\sigma^2_0}(\mu - \mu_0)^2 \right\} \](\( \mu_0, \sigma^2_0 \) は超パラメタ(hyperparameter)) 事後分布(まず \( \mu \) について式を整理する)\[ \begin{align} ~~~ p(\mu|\mathbf x) &\propto p(\mathbf x|\mu)p(\mu) \\ &\propto \exp\left\{ -\frac{1}{2\sigma^2}\sum_n(\mu - x_n)^2 - \frac{1}{2\sigma^2_0}(\mu - \mu_0)^2 \right\} \\ &= \exp \left\{ -\left( \frac{N}{2\sigma^2} + \frac{1}{2\sigma^2_0} \right)\mu^2 + 2\left( \frac{\sum_n x_n}{2\sigma^2} + \frac{\mu_0}{2\sigma^2_0} \right)\mu + \mathrm{const.} \right\} ~~~ \end{align} \] … 平方完成 \[ \therefore p(\mu|\mathbf x) = \mathcal N(\mu|\mu_N,\sigma^2_N) \]
ただし、
\[ \mu_N = \frac{\sigma^2}{N\sigma^2_0 + \sigma^2}\mu_0 + \frac{N\sigma^2_0}{N\sigma^2 + \sigma^2}\mu_\mathrm{ML},\ \frac{1}{\sigma^2_N} = \frac{1}{\sigma^2_0} + \frac{N}{\sigma^2} \](\( \mu_\mathrm{ML} \) は \( \mu \) の最尤推定解)
|
分散 \( \sigma^2 \) の推定(平均 \( \mu \) は既知)
(たとえば、リファレンス信号を入力してセンサーノイズの分散を測定する、というような場合)
|
尤度関数\[ \begin{align} p(\mathbf x|\lambda) &= \prod_{n=1}^N\mathcal N(x_n|\mu,\lambda^{-1}) \\ &= \prod_n\sqrt\frac{\lambda}{2\pi}\exp\left\{ -\frac{\lambda}{2}(x_n - \mu)^2 \right\} \\ &\propto \lambda^\frac{N}{2}\exp\left\{ -\frac{\sum_n(x_n - \mu)^2}{2}\lambda \right\} \end{align} \](精度 \( \lambda \equiv 1/\sigma^2 \)) 共役事前分布(ガンマ分布)\[ \begin{align} p(\lambda) &= \mathrm{Gam}(\lambda|a_0,b_0) = \frac{1}{\Gamma(a_0) }b_0^{a_0}\lambda^{a_0 - 1}\exp(-b_0\lambda) \\ &\propto \lambda^{a_0 - 1}\exp(-b_0\lambda) \end{align} \](\( a_0, b_0 \) はハイパーパラメタ) 事後分布\[ p(\lambda|\mathbf x) = p(\mathbf x|\lambda)p(\lambda) = \mathrm{Gam}(\lambda|a_N,b_N) \]ただし、\[ ~~~ a_N = a_0 + \frac{N}{2},\ b_N = b_0 + \sum_n\frac{(x_n - \mu)^2}{2} = b_0 + \frac{N}{2}\sigma^2_\mathrm{ML} ~~~ \](\( \sigma^2_\mathrm{ML} \) は \( \sigma^2 \) の最尤推定解) |
(「平均と分散の推定」については、PRMLに結論の式の記載なし)
共役事前分布
| もとの確率分布 | 推定したいパラメタ | 共役事前分布 |
|---|---|---|
| ベルヌーイ分布\[ \mathrm{Bern}(x|\mu) \] | \[ \mu \] | ベータ分布\[ \mathrm{Beta}(\mu|a,b) \] |
| 二項分布\[ \mathrm{Bin}(m|N,\mu) \] | \[ \mu \] | ベータ分布\[ \mathrm{Beta}(\mu|a,b) \] |
| ガウス分布\[ \mathcal N(x|\mu,\sigma^2) \] | \[ \mu \] | ガウス分布\[ \mathcal{N}(\mu|\mu_0,\sigma^2_0) \] |
| ガウス分布\[ \mathcal N(x|\mu,\lambda^{-1}) \] | \[ \lambda \] | ガンマ分布\[ \mathrm{Gam}(\lambda|a,b) \] |
| ガウス分布\[ \mathcal N(x|\mu,\lambda^{-1}) \] | \[ \mu, \lambda \] | ガウス–ガンマ分布\[\mathcal N(\mu|\mu_0,(\beta\lambda)^{-1})\mathrm{Gam}(\lambda|a,b) \] |
多次元ガウス分布のベイズ推定
データ \( \mathcal D = \{ 1.5, 1.3, 0.5, 0.6, 1.9 \} \) が、ガウス分布 \( p(x|\mu, 0.7^2) \) からそれぞれ独立にに得られたものとする(i.i.d. を仮定)
ベイズ推定の式(こちらの式の再掲載)\[ \begin{align} \mu_N &= \frac{\sigma^2}{N\sigma^2_0 + \sigma^2}\mu_0+\frac{N\sigma^2_0}{N\sigma_0^2 + \sigma^2}\mu_\mathrm{ML} \\ \frac{1}{\sigma^2_N} &= \frac{1}{\sigma^2_0} + \frac{N}{\sigma^2} \end{align} \]
データが一つずつ観測されたものとして、逐次推定(sequential estimation)を行う
\( N=1,\ \mu_\mathrm{ML} = \mathcal D_i \) とした式\[ \begin{align} \mu_{posterior} &= \frac{\sigma^2}{\sigma^2_{prior} + \sigma^2}\mu_{prior}+\frac{\sigma^2_{prior}}{\sigma_{prior}^2 + \sigma^2}D_i \\ \frac{1}{\sigma^2_{posterior}} &= \frac{1}{\sigma^2_{prior}} + \frac{1}{\sigma^2} \end{align} \](添え字 \( prior \) は事前確率分布のパラメタであること、\( posterior \) は事後確率分布のパラメタであることを示す)
分散は既知で \( \sigma^2 = 0.7^2 \)
事前分布のパラメタ(の初期値)は \( \mu_0 = 2,\ \sigma_0 = 0.5 \) とする(事前の信念)
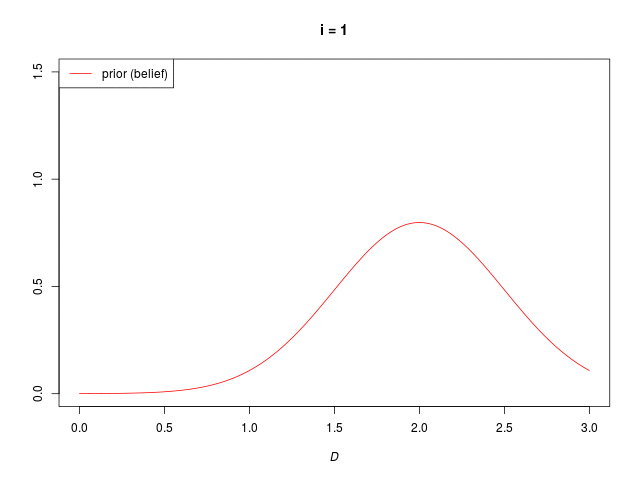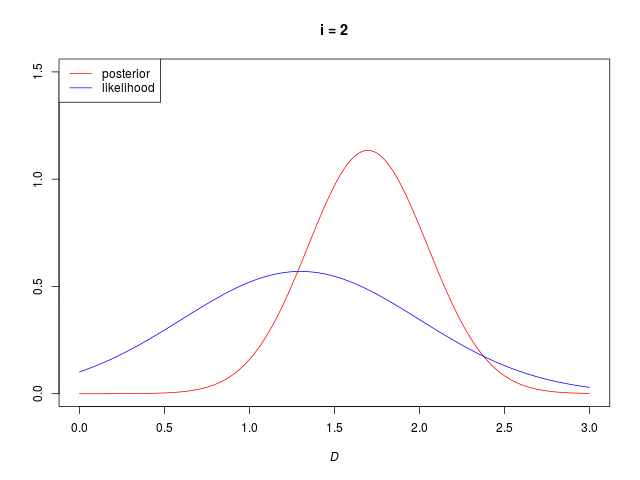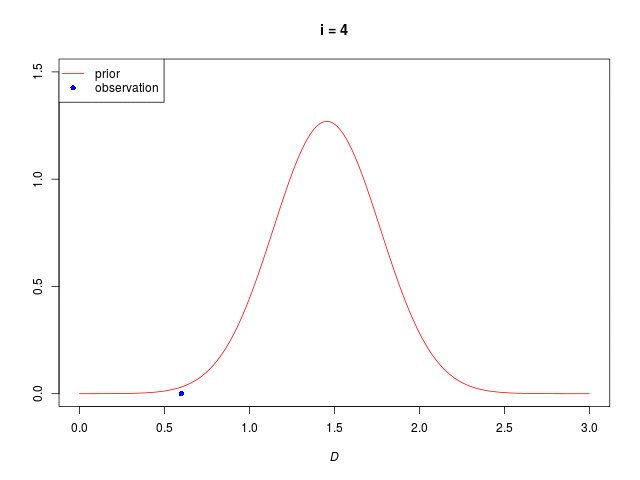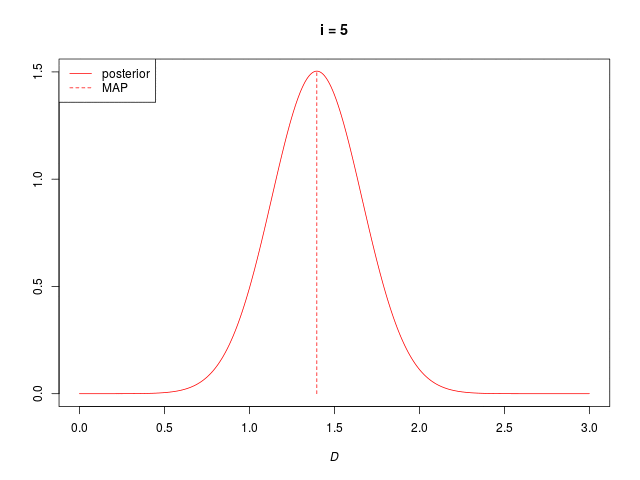
ソースコード
| R言語 |
|---|
|
\( N = 5 \) のデータをバッチ処理した結果と検算
プログラムで確認
| R言語 |
|---|
|
|
推定されたパラメタはどちらも \( \mu_N = 1.396552,\ \sigma_N = 0.2653343 \) で、逐次推定した推定結果とバッチ処理した推定結果は等しい
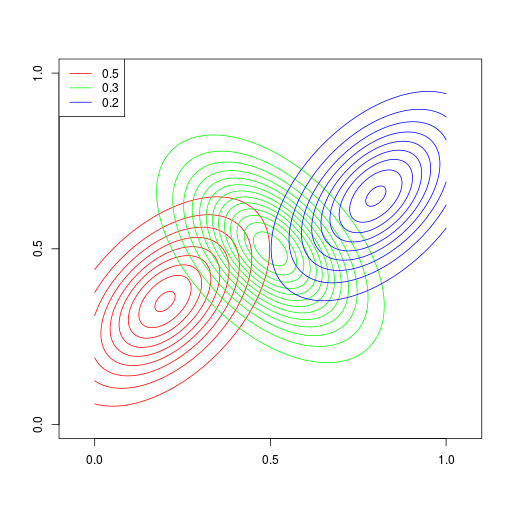
ガウス分布は単峰形(unimodal)である。ガウス分布のような基本的な分布をいくつか線形結合して、多峰形(multimodal)の分布を表現
 |  |  |
ソースコード
| R言語 |
|---|
|
|
ヒストグラム密度推定(histogram density estimation)

ソースコード
| R言語 |
|---|
|
R言語のdensity()関数
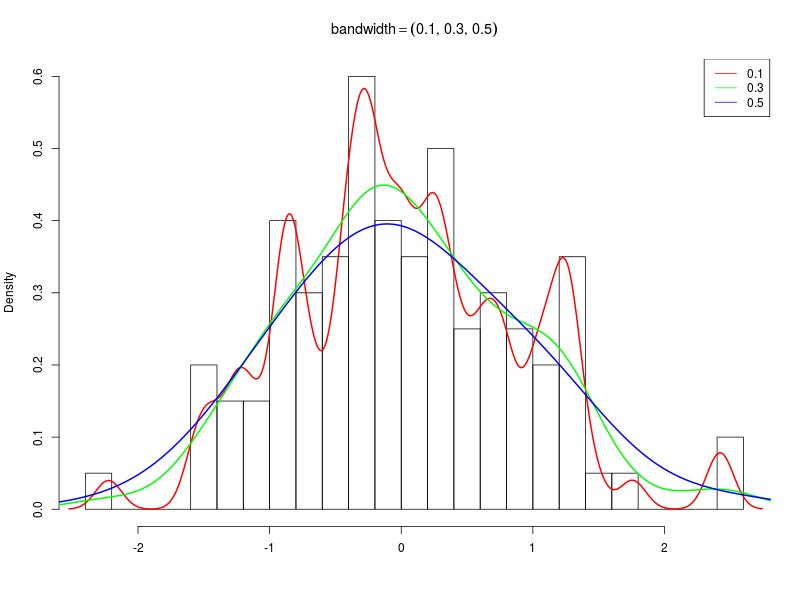
ソースコード
| R言語 |
|---|
|
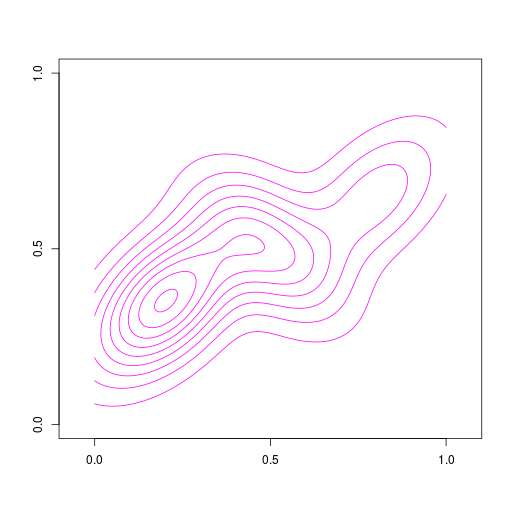
2次元のカーネル密度推定。R言語のMASSパッケージを利用
 | 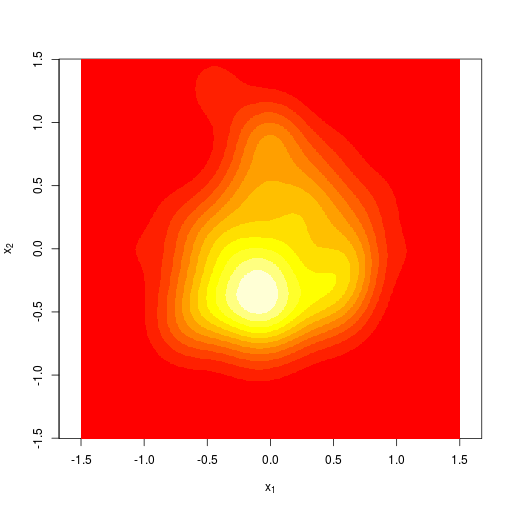 | 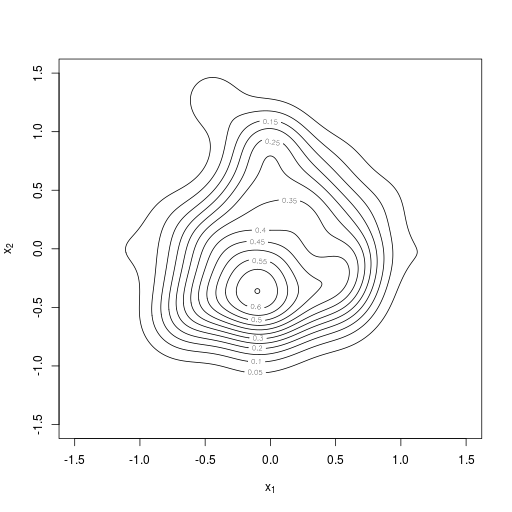 |
MASSパッケージのインストール(私の環境ではapt)
| Linux |
|---|
|
ソースコード
| R言語 |
|---|
|
目標変数(target variable) = 平均(多項式)+ ガウスノイズ\[ t = y(\mathbf x,\mathbf w) + \epsilon \]
\( \epsilon \) は、平均が 0 で精度 \( \beta \) のガウス分布の確率変数
確率の式として書く(\( \beta \) は精度で、分散の逆数(inverse variance))\[ p(t|\mathbf x, \mathbf w, \beta) = \mathcal N(t|y(\mathbf x, \mathbf w), \beta^{-1}) \]入力 \( \mathbf x \) を省略して\[ p(t|\mathbf w, \beta) = \mathcal N(t|y(\mathbf x, \mathbf w), \beta^{-1}) \]
\( x \) が与えられた \( t \) の条件付きガウス分布
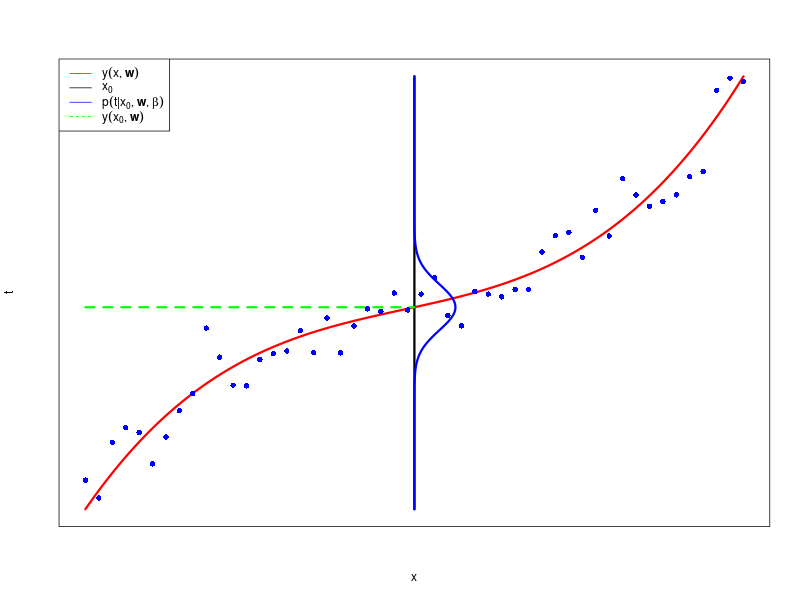
ソースコード
| R言語 |
|---|
|
入力変数の非線形な関数の線形結合を考える\[ y(\mathbf x, \mathbf w) = w_0 + \sum_{j=1}^{M-1}w_j\phi_j(\mathbf x) \]\( \phi_j(\mathbf x) \) は基底関数(basis function)
\( w_0 \) はバイアスパラメータ(bias parameter)。ダミー基底関数 \( \phi_0(\mathbf x) = 1 \)(恒等式)を導入\[ y(\mathbf x, \mathbf w) = \sum_{j=0}^{M-1}w_j\phi_j(\mathbf x) = \mathbf w^\mathrm T\boldsymbol\phi(\mathbf x) \]ただし、\( \mathbf w = (w_0, \ldots, w_{M-1})^\mathrm T,\ \boldsymbol\phi(\mathbf x) = \left( \phi_0(\mathbf x), \ldots, \phi_{M-1}(\mathbf x) \right)^\mathrm T \)
基底関数
| 多項式基底関数 | ガウス基底関数 |
|---|---|
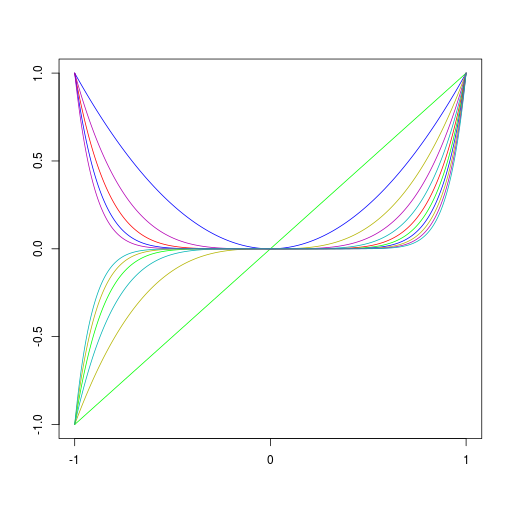 | 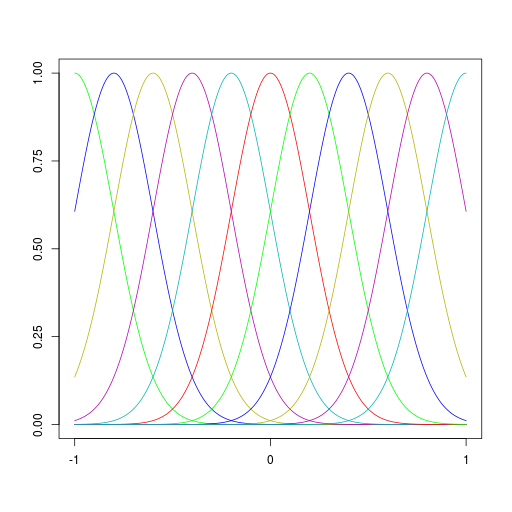 |
\[ \phi_j(x) = x^j \] | \[ \phi_j(x) = \exp\left\{ -\frac{(x - \mu_j)^2}{2s^2} \right\} \] |
ソースコード
| R言語 |
|---|
|
確率の式を線形基底関数を使って書く\[ p(t|\mathbf w, \beta) = \mathcal N(t|y(\mathbf x, \mathbf w), \beta^{-1}) = \mathcal N(t|\mathbf w^\mathrm T\boldsymbol \phi(\mathbf x), \beta^{-1}) \]
入力ベクトル \( \mathbf X = (\mathbf x_1, \ldots, \mathbf x_N)^\mathrm T \)、対応する目標ベクトル(target vector) \( \mathbf t = (t_1, \ldots, t_N)^\mathrm T \) からなる訓練データ集合(\( N \) 回の観測)を考える
尤度関数\[ p(\mathbf t|\mathbf X, \mathbf w, \beta) = \prod_{n=1}^{N}\mathcal N(t_n|\mathbf w^\mathrm T\boldsymbol\phi(\mathbf x_n), \beta^{-1}) \](i.i.d.を仮定しているので、積の形で書ける)
\( \mathbf X \) を省略して\[ p(\mathbf t|\mathbf w, \beta) = \prod_{n=1}^{N}\mathcal N(t_n|\mathbf w^\mathrm T\boldsymbol\phi(\mathbf x_n), \beta^{-1}) \]
対数尤度関数\[ \begin{align} \ln p(\mathbf t|\mathbf w,\beta) &= \sum_{n=1}^N\ln\mathcal N(t_n|\mathbf w^\mathrm T\boldsymbol\phi(\mathbf x_n),\beta^{-1}) \\ &= \frac{N}{2}\ln\beta - \frac{N}{2}\ln(2\pi) - \frac{\beta}{2}\sum_{n=1}^N\left\{ t_n - \mathbf w^\mathrm T\boldsymbol\phi(\mathbf x_n) \right\}^2 \end{align} \](対数をとると積が和になる)
\( \mathbf w \) について対数尤度を最大化する
|
\( \mathbf w\) について偏微分して \( 0 \) になるところ\[ \nabla\ln p(\mathbf t|\mathbf w, \beta) = \beta\sum_{n=1}^N\left\{t_n-\mathbf w^\mathrm T\boldsymbol\phi(\mathbf x_n)\right\}\boldsymbol\phi(\mathbf x_n) = 0 \](\( \mathbf w \) の無い項は消える。2次式の微分 ) 解き方\[ \begin{align} \beta\sum_{n=1}^N\left\{ t_n - \mathbf w^\mathrm T\boldsymbol\phi(\mathbf x_n) \right\}\boldsymbol\phi(\mathbf x_n) &= 0 \\ \sum_{n=1}^N\left\{ t_n - \mathbf w^\mathrm T\boldsymbol\phi(\mathbf x_n) \right\}\boldsymbol\phi(\mathbf x_n) &= 0 \\ \sum_{n=1}^Nt_n\boldsymbol\phi(\mathbf x_n) &= \sum_{n=1}^N\left\{ \mathbf w^\mathrm T\boldsymbol\phi(\mathbf x_n) \right\}\boldsymbol\phi(\mathbf x_n) \end{align} \](\( \beta \) は消すことができる) \( \sum \) 記号を行列(とベクトル)で書く\[ \begin{align} \sum_{n=1}^Nt_n\boldsymbol\phi(\mathbf x_n) &= \sum_{n=1}^N\left( \mathbf w^\mathrm T\boldsymbol\phi(\mathbf x_n) \right)\boldsymbol\phi(\mathbf x_n) \\ ~~~ \begin{pmatrix} \phi_0(\mathbf x_1) & \cdots & \phi_0(\mathbf x_N) \\ \vdots & \ddots & \vdots \\ \phi_{M-1}(\mathbf x_1) & \cdots & \phi_{M-1}(\mathbf x_N) \end{pmatrix}\begin{pmatrix} t_1 \\ \vdots \\ t_n \end{pmatrix} &= \begin{pmatrix} \phi_0(\mathbf x_1) & \cdots & \phi_0(\mathbf x_N) \\ \vdots & \ddots & \vdots \\ \phi_{M-1}(\mathbf x_1) & \cdots & \phi_{M-1}(\mathbf x_N) \end{pmatrix}\begin{pmatrix} \mathbf w^\mathrm T\boldsymbol{\phi}(\mathbf x_1) \\ \vdots \\ \mathbf w^\mathrm T\boldsymbol{\phi}(\mathbf x_N) \end{pmatrix} ~~~ \\ &= \begin{pmatrix} \phi_0(\mathbf x_1) & \cdots & \phi_0(\mathbf x_N) \\ \vdots & \ddots & \vdots \\ \phi_{M-1}(\mathbf x_1) & \cdots & \phi_{M-1}(\mathbf x_N) \end{pmatrix}\begin{pmatrix} \boldsymbol{\phi}(\mathbf x_1)^\mathrm T \\ \vdots \\ \boldsymbol{\phi}(\mathbf x_N)^\mathrm T \end{pmatrix}\mathbf w \end{align} \](基底が \( M \) 次元、観測データ(の個数)が \( N \) 次元) (転置されていたりするが、同じ形の行列が何度も出てくるので名前を付ける。この(↓)形を)計画行列(design matrix) \( \boldsymbol \Phi \) とする\[ \boldsymbol\Phi = \begin{pmatrix} \boldsymbol\phi(\mathbf x_1)^\mathrm T \\ \vdots \\ \boldsymbol\phi(\mathbf x_N)^\mathrm T \end{pmatrix} = \begin{pmatrix} \phi_0(\mathbf x_1) & \cdots & \phi_{M-1}(\mathbf x_1) \\ \vdots & \ddots & \vdots \\ \phi_0(\mathbf x_N) & \cdots & \phi_{M-1}(\mathbf x_N) \end{pmatrix} \](計画行列は \( N\times M \) 行列。\( N \) は観測の数、\( M \) は基底関数の次元数) 計画行列を用いる\[ \begin{align} \sum_{n=1}^Nt_n\boldsymbol\phi(\mathbf x_n) &= \sum_{n=1}^N\left( \mathbf w^\mathrm T\boldsymbol\phi(\mathbf x_n) \right)\boldsymbol\phi(\mathbf x_n) \\ ~~~ \begin{pmatrix} \phi_0(\mathbf x_1) & \cdots & \phi_0(\mathbf x_N) \\ \vdots & \ddots & \vdots \\ \phi_{M-1}(\mathbf x_1) & \cdots & \phi_{M-1}(\mathbf x_N) \end{pmatrix}\begin{pmatrix} t_1 \\ \vdots \\ t_n \end{pmatrix} &= \begin{pmatrix} \phi_0(\mathbf x_1) & \cdots & \phi_0(\mathbf x_N) \\ \vdots & \ddots & \vdots \\ \phi_{M-1}(\mathbf x_1) & \cdots & \phi_{M-1}(\mathbf x_N) \end{pmatrix}\begin{pmatrix} \boldsymbol{\phi}(\mathbf x_1)^\mathrm T \\ \vdots \\ \boldsymbol{\phi}(\mathbf x_N)^\mathrm T \end{pmatrix}\mathbf w ~~~ \\ \boldsymbol\Phi^\mathrm T\mathbf t &= \boldsymbol\Phi^\mathrm T\boldsymbol\Phi\mathbf w \end{align} \](\( \mathbf t \) は目標ベクトル、\( \mathbf w \) は係数ベクトル) パラメタ \( \mathbf w \) について解く(\( \mathbf w \) の最尤推定解)\[ \therefore \mathbf w_\mathrm{ML} = \left( \boldsymbol\Phi^\mathrm T\boldsymbol\Phi \right)^{-1}\boldsymbol\Phi^\mathrm T\mathbf t = \boldsymbol\Phi^\dagger\mathbf t \](\( \boldsymbol\Phi^\dagger \) は計画行列の疑似逆行列。行列が退化(ランク落ち)してなければ求まる) |
\( \beta \) について対数尤度を最大化する
|
対数尤度関数\[ ~~~ \ln p(\mathbf t|\mathbf w,\beta) = \frac{N}{2}\ln\beta - \frac{N}{2}\ln(2\pi) - \frac{\beta}{2}\sum_{n=1}^N\left\{ t_n - \mathbf w^\mathrm T\boldsymbol\phi(\mathbf x_n) \right\}^2 ~~~ \] \( \beta \) について偏微分して \( 0 \) になるところ\[ \frac{\partial}{\partial\beta}\ln p(\mathbf t|\mathbf w, \beta) = \frac{N}{2\beta} - \frac{1}{2}\sum_{n=1}{N}\left\{ t_n - \mathbf w^\mathrm T\boldsymbol \phi(\mathbf x_n) \right\}^2 = 0 \] \( \beta \) の最尤推定解\[ \therefore \frac{1}{\beta_\mathrm{ML}} = \frac{1}{N}\sum_{n=1}^N\left\{ t_n - \mathbf w_\mathrm{ML}^\mathrm T\boldsymbol\phi(\mathbf x_n) \right\}^2 \] 計画行列を用いて書けば\[ \therefore \frac{1}{\beta_\mathrm{ML}} = \frac{1}{N}(\boldsymbol\Phi\mathbf w_\mathrm{ML} - \mathbf t)^\mathrm T(\boldsymbol\Phi\mathbf w_\mathrm{ML} - \mathbf t) \] |
多項式基底関数(polynomial basis function)の例。基底関数は \( \boldsymbol\phi(x) = (1, x, x^2, x^3)^\mathrm T \)
スクリプトと計算結果
| Octave | R言語 |
|---|---|
|
|
|
|
データセットは上記と同じものを使用。最尤解 \( \mathbf w_\mathrm{ML} \) が、最小二乗法で求めた解に一致することを確認
\( \mathbf w \) の事後分布 ← 尤度関数 \( \mathbf w_\mathrm{ML} \) × \( \mathbf w \) の事前分布
\( \beta \) は既知とする
|
尤度関数\[ \begin{align} p(\mathbf{t}|\mathbf{w}) &= \prod_{n=1}^N\mathcal{N}(t_n|\mathbf{w}^\mathrm{T}\boldsymbol\phi(\mathbf x_n),\beta^{-1}) \\ &= \prod_{n=1}^N\frac{1}{\sqrt{2\pi\beta^{-1}}}\exp\left\{ -\frac{\beta}{2}\left(t_n - \mathbf{w}^\mathrm{T}\boldsymbol\phi(\mathbf x_n)\right)^2 \right\} \\ &= \frac{1}{(2\pi\beta^{-1})^{N/2}}\exp\left\{ -\frac{\beta}{2}\sum_{n=1}^N\left(t_n - \mathbf{w}^\mathrm{T}\boldsymbol\phi(\mathbf x_n)\right)^2 \right\} \\ &= \frac{1}{(2\pi\beta^{-1})^{N/2}}\exp\left\{ -\frac{\beta}{2}(\mathbf{t} - \mathbf{\Phi w})^\mathrm{T}(\mathbf{t} - \mathbf{\Phi w}) \right\} \\ &= \mathcal{N}(\mathbf{t}|\mathbf{\Phi w},\beta^{-1}\mathbf{I}) \end{align} \](1次元のガウス分布のかけ算が、一つの多次元ガウス分布として書くことができる) 事前分布\[ p(\mathbf{w}) = \mathcal{N}(\mathbf{w}|\mathbf{m}_0,\mathbf{S}_0) \](\( \mathbf m_0 \) と \( \mathbf S_0 \)(分布の中心と共分散)は事前の信念に従って適当に決める) 考えているモデル\[ \begin{align} p(\mathbf{w}) &= \mathcal{N}(\mathbf{w}|\mathbf{m}_0,\mathbf{S}_0) \\ p(\mathbf{t}|\mathbf{w}) &= \mathcal{N}(\mathbf{t}|\mathbf{\Phi w},\beta^{-1}\mathbf{I}) \end{align} \]に、ガウス分布の公式をあてはめる 事後分布\[ p(\mathbf w|\mathbf t)=\mathcal{N}(\mathbf{w}|\mathbf{m}_N,\mathbf{S}_N) \](\( \mathbf m_N \) と \( \mathbf S_N \) は \( N \) 個のデータを観測した後に(ベイズ推定によって)更新された新たな信念) パラメタ \( \mathbf m_N \) と \( \mathbf S_N \)\[ \begin{align} \mathbf{m}_N &= \mathbf{S}_N\left(\mathbf{S}_0^{-1}\mathbf{m}_0 + \beta\boldsymbol{\Phi}^\mathrm{T}\mathbf{t}\right) \\ \mathbf{S}_N^{-1} &= \mathbf{S}_0^{-1} + \beta\boldsymbol{\Phi}^\mathrm{T}\boldsymbol{\Phi} \end{align} \] 事前分布が \( 0 \) 中心で等方的なガウス分布の場合\[ \begin{align} \mathbf{m}_0 &= \mathbf{0} \\ \mathbf{S}_0 &= \alpha^{-1}\mathbf{I} \end{align} \]\[ \begin{align} \mathbf{m}_N &= \beta\mathbf{S}_N\boldsymbol\Phi^\mathrm{T}\mathbf{t} \\ \mathbf{S}_N^{-1} &= \alpha\mathbf{I} + \beta\boldsymbol\Phi^\mathrm{T}\boldsymbol\Phi \end{align} \](ハイパーパラメータは \( \alpha \)、\( \beta \)(、パラメータは \( \mathbf w \))) |
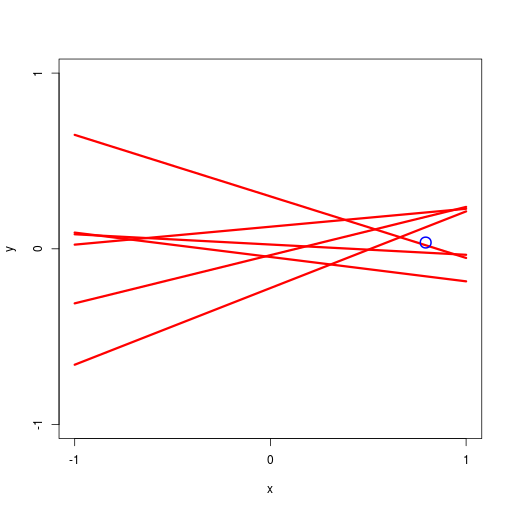
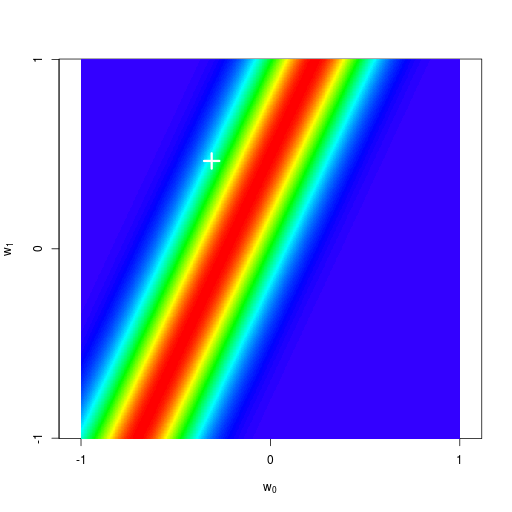
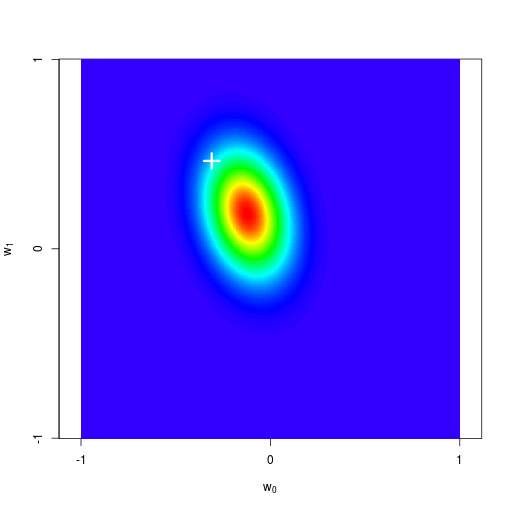
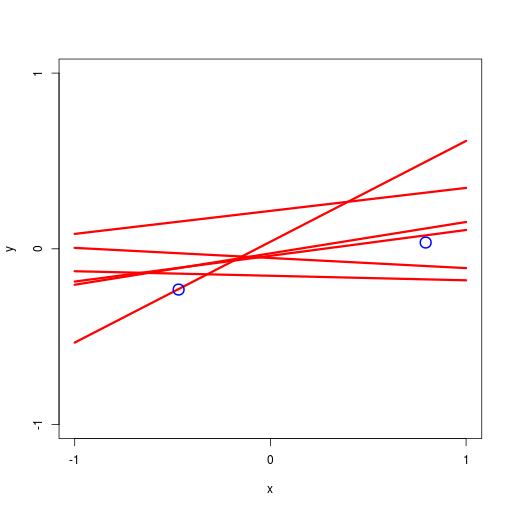
逐次ベイズ学習(sequential Bayesian learning)の例
| prior | data space | |
|---|---|---|
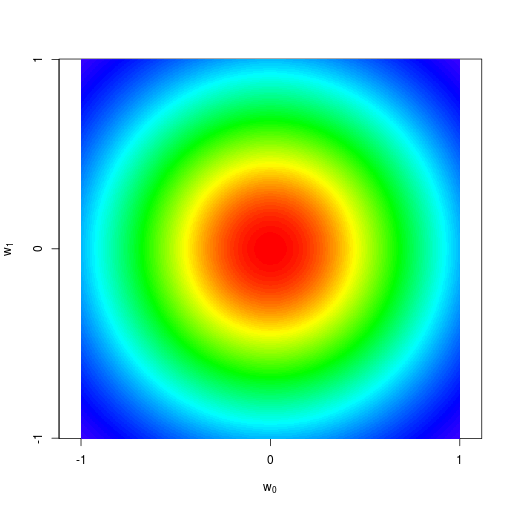 |
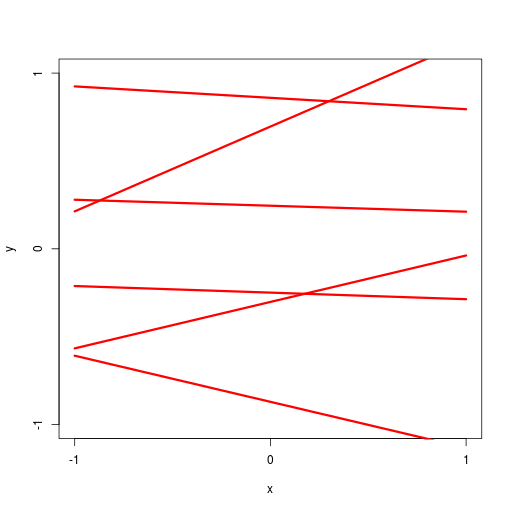 |
|
| likelihood | prior/posterior | data space |
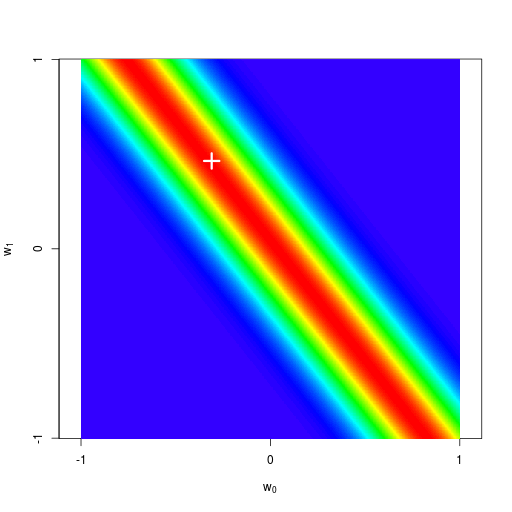 |
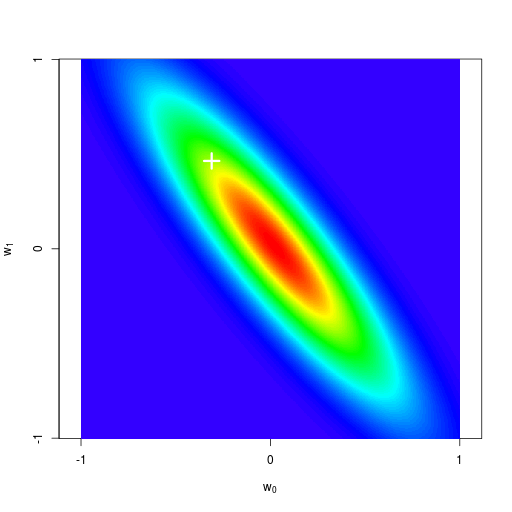 |
 |
 |
 |
 |
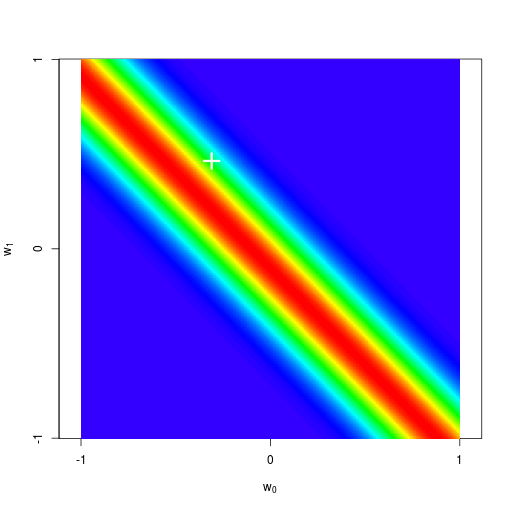 |
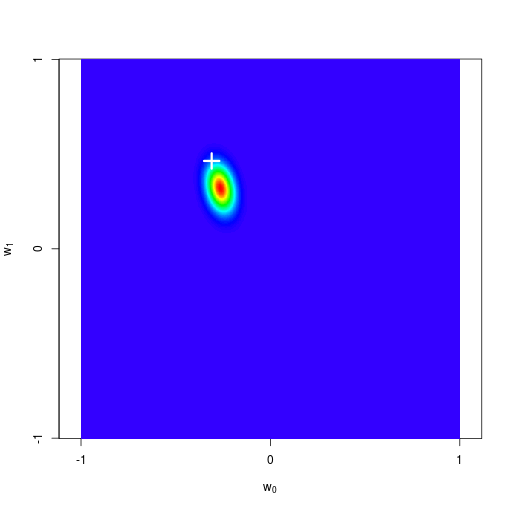 |
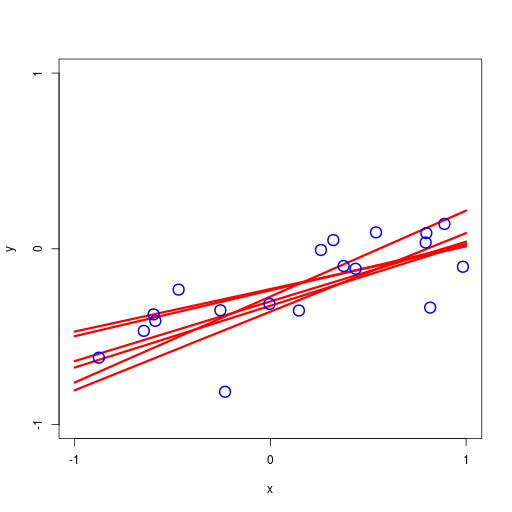 |
ソースコード
| R言語 |
|---|
|
実際には、通常 \( \mathbf w \) の値そのものには興味がなく、むしろ新しい \( \mathbf x\) の値に対する \( t \) を予測したい。そのためには、\[ p(t|\mathbf t) = \int p(t|\mathbf w)p(\mathbf w|\mathbf t)\,\mathrm d\mathbf w = \int\mathcal N(t|\mathbf w^\mathrm T\boldsymbol\phi(\mathbf x), \beta^{-1})\mathcal N(\mathbf w|\mathbf m_N, \mathbf S_N)\,\mathrm d\mathbf w \]で定義される予測分布を評価する必要がある
|
計算。予測分布\[ p(t|\mathbf t) = \int p(t|\mathbf w) p(\mathbf w|\mathbf t)\,\mathrm d\mathbf w \]\[ p(t|\mathbf w) = \mathcal N(t|\mathbf w^\mathrm T \boldsymbol\phi(\mathbf x), \beta^{-1}),\ p(\mathbf w|\mathbf t) = \mathcal N(\mathbf w|\mathbf m_N, \mathbf S_N) \] ガウス分布の公式をあてはめる\[ \therefore p(t|\mathbf t) = \mathcal N(t|\mathbf m_N^\mathrm T \boldsymbol\phi(\mathbf x), \frac{1}{\beta} + \boldsymbol\phi(\mathbf x)^\mathrm T \mathbf S_N \boldsymbol\phi(\mathbf x)) \] 予測分布の平均 \( \mathbf m_N^\mathrm T \boldsymbol\phi(\mathbf x) \) は、MAP解に一致している(\( \mathbf w_\mathrm{MAP} = \mathbf m_N \)) 予測分布の分散は、既知とした精度 \( \beta \) の逆数(分散)に、\( \boldsymbol\phi(\mathbf x)^\mathrm T \mathbf S_N \boldsymbol\phi(\mathbf x) \) の項だけ足したものになっている(分散がその分だけ大きくなっている) | |
| cf. |
ベイズ推定の結果のMAP解\[ p(\mathbf w|\mathbf t)=\mathcal N(\mathbf w|\mathbf m_N, \mathbf S_N),\ \mathbf w_\mathrm{MAP} = \mathbf m_N \] 線形基底関数モデルの式\[ p(t|\mathbf w, \beta) = \mathcal N(t|\mathbf w^\mathrm T \boldsymbol\phi(\mathbf x), \beta^{-1}) \]\( \mathbf w \) にMAP解 \( \mathbf w_\mathrm{MAP} \) を採用\[ p(t|\mathbf w_\mathrm{MAP}, \beta) = \mathcal N(t|\mathbf w_\mathrm{MAP}^\mathrm T \boldsymbol\phi(\mathbf x), \beta^{-1}) = \mathcal N(t|\mathbf m_N^\mathrm T \boldsymbol\phi(\mathbf x), \beta^{-1}) \] |
|---|---|
予測分布の例
 |  |
ソースコード
| R言語 |
|---|
|
モデル選択、モデルエビデンスなど
《積み残し》
© 2020 Tadakazu Nagai